Schweres akutes Atemwegssyndrom
| Klassifikation nach ICD-10 | |
|---|---|
| U04.9 | Schweres akutes respiratorisches Syndrom [SARS], nicht näher bezeichnet |
| J17.1* | Pneumonie bei anderenorts klassifizierten Viruskrankheiten |
| ICD-10 online (WHO-Version 2019) | |
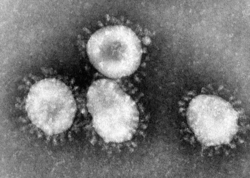
Das schwere akute Atemwegssyndrom, auch Schweres Akutes Respiratorisches Syndrom (englisch severe acute respiratory syndrome, SARS) genannt, ist eine Schwere Akute Atemwegsinfektion (SARI), die erstmals im November 2002 in der südchinesischen Provinz Guangdong beobachtet wurde.[1] Laut dem Bernhard-Nocht-Institut für Tropenmedizin in Hamburg entspricht das klinische Bild einer atypischen Lungenentzündung (Pneumonie). Der Erreger von SARS war ein bis dahin unbekanntes Coronavirus, das man inzwischen als „SARS-Coronavirus“ (SARS-CoV-1) bezeichnet. Die SARS-Pandemie 2002/2003 forderte knapp 800 Todesopfer.[2]
Erreger

Entdeckung
Bakterielle Erreger wie Chlamydien, Mykoplasmen oder Legionellen, welche normalerweise eine atypische Lungenentzündung verursachen, wurden bei dieser neuen Form nicht gefunden. Da die Erkrankten auf eine Behandlung mit Antibiotika nicht ansprachen, lag die Vermutung nahe, dass der Erreger ein Virus ist.
Zunächst wurden Paramyxoviren als Ursache von SARS vermutet. Um den 26. März 2003 erhärtete sich jedoch der Verdacht, dass der Erreger zur Familie Coronaviridae gehört. Er wurde gleichzeitig an der Universität Hongkong, an den Centers for Disease Control and Prevention (CDC) in Atlanta und am Bernhard-Nocht-Institut für Tropenmedizin in Hamburg als neues Coronavirus identifiziert. Anhand der Gensequenzen wurde vermutet, dass ein bekanntes Coronavirus entweder mutiert oder dass eine Virusart, die bisher nur Tiere befallen hat, auf den Menschen „übergesprungen“ war. Weitere Untersuchungen konzentrierten sich daher auf das neu entdeckte Coronavirus und als weitere Faktoren auf Chlamydien und Paramyxoviren. Bisher sei nichts gefunden worden, „was gegen die ursächliche Rolle des Coronavirus spricht“, hieß es aus Fachkreisen. Am 12. April 2003 gab Marco Marra, Direktor des Michael Smith Genome Sciences Centre der British Columbia Cancer Agency, bekannt, es sei ihm gemeinsam mit anderen kanadischen Forschern gelungen, die RNA-Sequenz des Virus zu entschlüsseln. Vier Tage darauf veröffentlichte die WHO eine Pressemitteilung mit der Erklärung, das von mehreren Laboratorien identifizierte Coronavirus sei der offizielle Auslöser von SARS. Es erhielt die wissenschaftliche Bezeichnung „severe acute respiratory syndrome coronavirus“ (SARS-CoV). Zwei Tage nach Identifizierung des Virus gelang es den Virologen Christian Drosten und Stephan Günther vom Bernhard-Nocht-Institut als ersten, einen diagnostischen Test zu entwickeln, der inzwischen weltweit eingesetzt wird.[3] Die Gruppen vom CDC, der Erasmus-Universität in Rotterdam, der University of San Francisco und Bernhard-Nocht-Institut publizierten gemeinsam[4] 2003 in Science über die Entdeckung des SARS-Coronavirus und der Bestimmung von dessen Genomsequenz ebenso wie die kanadische Gruppe um Marco Marra.[5]
Abstammung
Einige Experten vermuteten zunächst, das Virus hinter der asiatischen Lungenkrankheit könnte von seltenen wilden Tieren als Hauptwirt (Reservoirwirt) (z. B. den Larvenrollern (Paguma larvata) aus der Familie der Schleichkatzen) stammen, die in Südchina gerne als Delikatesse verzehrt werden. Letzteres wird durch Berichte in der singapurchinesischen Tageszeitung Lianhe Wanbao unterstützt, wonach die Krankheit vom Koch eines Spezialitätenrestaurants für wilde Tiere in Shenzhen in Südchina ausgegangen sein könnte. Auch Virologen eines WHO-Teams halten diese Theorie für plausibel. Für die mechanische Übertragung der Viren sind Kakerlaken als Vektor festgestellt worden.
Im September 2005 ergab eine Studie an verschiedenen wildlebenden Tieren im Hongkonger Großraum, dass das SARS-Coronavirus auch von der Chinesischen Hufeisennase (Rhinolophus sinicus) aus der Familie der Hufeisennasen (Rhinolophidae) – einer Familie innerhalb der Ordnung der Fledertiere (Fledermäuse) – übertragen wird. In 40 Prozent der Abstriche wurden Coronaviren gefunden, die genetisch große Ähnlichkeit mit dem beim Menschen und bei Larvenrollern identifizierten SARS-Erreger besitzen. Die Fledermäuse zeigten keine Krankheitsanzeichen. 2013 und 2017 wurden weitere genetische Hinweise publiziert, die auf Chinesische Hufeisennasen als Reservoirwirt verweisen.[6][7] Es ist jedoch weiterhin nicht zweifelsfrei geklärt, ob Fledermäuse oder Schleichkatzen der ursprüngliche Reservoirwirt des Virus sind. Die chinesischen Fledermäuse könnten auch ein direkter Übertragungsvektor auf den Menschen sein, da sie ebenso wie Schleichkatzen als Delikatesse gelten und ihr Kot in der traditionellen chinesischen Medizin (TCM) verwendet wird.
Übertragung
Die ursprüngliche Annahme, dass das Virus an der Luft nicht sehr resistent sei, musste ebenfalls revidiert werden. Es überlebt nach neueren Erkenntnissen bis zu 24 Stunden außerhalb des menschlichen Körpers. Eine Übertragung über Klimaanlagen hält man dennoch für nicht sehr wahrscheinlich.
Die Erregerübertragung erfolgt wohl überwiegend durch Tröpfcheninfektion aus kurzer Distanz und damit bei Kontakt mit hustenden und niesenden Infizierten. Der indirekte Weg über Kontaktinfektion bzw. Schmierinfektion mit den Viren der auf Gegenständen oder Körperoberflächen niedergegangenen infektiösen Exspirationströpfchen, wenn sie anschließend über die Schleimhäute z. B. in Mund, Nase oder Augen in den Körper gelangen, kann nicht ausgeschlossen werden.[8] Eine Übertragung über Körperausscheidungen (den fäko-oralen Weg) kann ebenfalls nicht ausgeschlossen werden. Weiterhin sei laut WHO eine Erregerübertragung durch infizierte Tiere (z. B. Kakerlaken) möglich.

Symptome
Die Inkubationszeit beträgt zwei bis sieben Tage. Symptome sind laut der Weltgesundheitsorganisation:
- plötzlich auftretendes, schnell steigendes, hohes Fieber (über 38 °C)
- Halsentzündung mit Husten und Heiserkeit
- Atemnot
- Muskelschmerzen
- Kopfschmerzen
- Entzündung beider Lungenflügel
Ersten Laboruntersuchungen zufolge ist es möglich, dass auch die Abnahme der Zahl der Blutplättchen (Thrombozytopenie) und der weißen Blutkörperchen (Leukozytopenie) eine Folge der Krankheit ist.
Therapie
Spezielle Behandlungsmaßnahmen gibt es noch nicht. Ärzte verabreichten damals zunächst als Virostatikum das Nukleosid-Analogon Ribavirin, das auch bei Hepatitis C eingesetzt wird, sowie Cortison. Danach erhielten die Betroffenen meist eine Mischung aus verschiedenen Antibiotika, um die begleitende Entzündung der Atemwege durch Bakterien abheilen zu lassen, denn die zusätzliche bakterielle Infektion macht die Lungenentzündung erst so gefährlich. Die RNA-Sequenz des mutmaßlichen SARS-Erregers ist zwar später in Kanada entschlüsselt worden, jedoch ist die Entwicklung eines wirksamen Medikaments noch nicht in Sicht. Mit Hilfe der Gen-Daten könnten zwar neue Diagnoseverfahren entwickelt werden; aber es wird noch eine unbestimmte Zeit brauchen, bis Arzneimittel oder Impfstoffe zur Verfügung stehen.
Impfstoffentwicklung
Das US-amerikanische Forscherteam um Hilary Koprowski veränderte Anfang 2005 Tomaten- und Tabakpflanzen genetisch so, dass die Pflanzen einen Teil des so genannten Spike-Glykoproteins des SARS-Virus produzieren. Dieses so erhaltene Protein, das sich normalerweise in der Außenhülle des Virus befindet und dem Erreger hilft, in die menschlichen Zellen einzudringen, wurde nun als ein Impfstoff gegen das Virus bei Mäusen erfolgreich getestet. Weitere Studien hinsichtlich der besseren Verabreichung des Impfstoffes sind jedoch noch notwendig.[9]
Nachbeobachtung der Patienten und Antikörperbildung
Rund 25 % der SARS-Patienten, welche an einem Militärkrankenhaus in Peking nach Entlassung betreut wurden, zeigten verbleibende Lungenstrukturveränderung im Sinne einer Lungenfibrose. 40 Patienten hatten eine messbare Diffusionskapazitätsstörung der Lunge. Diese besserte sich spontan bei der Hälfte der Betroffenen. Auch die radiologisch dargestellte Lungenfibrose zeigte im einjährigen Nachbehandlungszeitraum eine Besserungstendenz, verschwand aber nicht bei allen Patienten vollständig. Die Autoren stellten die Hypothese auf, dass aufgrund der Besserung ein anderer Mechanismus als bei gewöhnlichen Lungenfibrosen bestünde. In derselben Studie zeigten sich IgG-Antikörper gegen das Virus bei rund 80 % der Patienten. Aufgrund der Veränderung der Antikörpertiter im Verlauf gehen die Autoren nur von einer zeitlich begrenzten Immunität aus.[10]
Im Rahmen einer sehr kleinen Langzeit-Studie wurden infizierte Mitarbeiter des Gesundheitswesens als SARS-Patienten über 15 Jahre beobachtet. Dabei zeigte sich eine Besserung der Lungenschädigung der Patienten, welche bei Abheilung radiologisch nachweisbare Residuen der Erkrankung hatten. Nach einem Jahr stellte sich jedoch eine Plateauphase mit milden restriktiven Störungen und Diffusionsstörungen ein. Die in den früheren Nachbehandlungsuntersuchungen beobachtete Femurkopfnekrose wurde als Spätfolge der Steroidtherapie im Rahmen der Behandlung des SARS gesehen.[11]
Warnungen
Am 12. März 2003 stufte die Weltgesundheitsorganisation (WHO) SARS als weltweite Bedrohung ein.[12]
Meldepflicht
Im Deutschland besteht für den Erreger Severe-Acute-Respiratory-Syndrome-Coronavirus (SARS-CoV) eine namentliche Meldepflicht laut § 7 Infektionsschutzgesetz (IfSG). Es muss der direkte und indirekte Nachweis gemeldet werden, soweit die Nachweise auf eine akute Infektion hinweisen. Im Regelfall besteht diese Verpflichtung für die Leitung des untersuchenden Labors (siehe § 8 IfSG).
In Österreich ist SARS (Schweres Akutes Respiratorisches Syndrom) eine anzeigepflichtige Krankheit gemäß § 1 Abs. 1 Nr. 1 Epidemiegesetz 1950. Die Anzeigepflicht bezieht sich auf Verdachts-, Erkrankungs- und Todesfälle. Zur Anzeige verpflichtet sind unter anderen Ärzte und Labore (§ 3 Epidemiegesetz 1950).
In der Schweiz besteht Meldepflicht für SARS in Bezug auf klinischen Verdacht, Rücksprache mit Fachärztin oder Facharzt für Infektiologie. Veranlassung einer erregerspezifischen Labordiagnostik und epidemiologischen Zusammenhang durch den behandelnden Arzt. Zudem bei positiven und negativen Laborbefund für den Erreger SARS-Coronavirus durch das untersuchende Labor. Dies ergibt sich aus dem Epidemiengesetz (EpG) in Verbindung mit der Epidemienverordnung und Anhang 1 bzw. Anhang 3 der Verordnung des EDI über die Meldung von Beobachtungen übertragbarer Krankheiten des Menschen.
Literatur
- I. Stock: Coronaviren: Erreger von SARS und anderen Infektionen. In: Medizinische Monatsschrift für Pharmazeuten, 2004, 27/1, S. 4–12, ISSN 0342-9601.
- S. Hecker: SARS und Vogelgrippe – Die Wissenslücken. In: Österreichische Ärztezeitung, 4/2004, S. 30–31, ISSN 0029-8786.
- R. Gottschalk: Neue und hochinfektiöse Krankheitserreger – Seuchenschutz durch den Öffentlichen Gesundheitsdienst am Beispiel SARS. Akademie für Öffentliches Gesundheitswesen, Düsseldorf 2005, ISBN 3-9807313-5-9.
- Michael Schillmeier, Wiebke Pohler: Kosmo-politische Ereignisse. Zur sozialen Topologie von SARS. In: Soziale Welt, 2006, Heft 4, S. 331–349.
Weblinks
- Schweres Akutes Respiratorisches Syndrom (SARS) – Informationen des Robert Koch-Instituts
- SARS (Schweres Akutes Respiratorisches Syndrom) aus: Infektionskrankheiten A–Z und SARS: Informationen für medizinisches Fachpersonal (PDF) jeweils vom Bundesministerium für Soziales, Gesundheit, Pflege und Konsumentenschutz (Österreich)
- SARS 2003/04 weltweit. Bundesamt für Gesundheit (Schweiz)
- Informationen zu SARS. WHO
- Special Online Collection – The SARS Epidemic (Version vom 25. April 2009 im Internet Archive; Originalseite nicht mehr existent) Science Magazine.
Einzelnachweise
- ↑ Barbara Bertisch: Kleine Influenza-Historie. 2009
- ↑ SARS-Fälle. WHO (englisch)
- ↑ Virologen des Tropeninstituts erhalten Preis der Werner Otto Stiftung. Bernhard-Nocht-Institut für Tropenmedizin, 4. Dezember 2003, archiviert vom (nicht mehr online verfügbar) am 16. März 2020; abgerufen am 21. Januar 2020. Info: Der Archivlink wurde automatisch eingesetzt und noch nicht geprüft. Bitte prüfe Original- und Archivlink gemäß Anleitung und entferne dann diesen Hinweis.
- ↑ Paul A. Rota, William J. Bellini, Christian Drosten, Stephan Günther, Albert Osterhaus, Joseph DeRisi u. a.: Characterization of a Novel Coronavirus Associated with Severe Acute Respiratory Syndrome. In: Science. Band 300, Nr. 5624, 30. Mai 2003, ISSN 0036-8075, S. 1394–1399, doi:10.1126/science.1085952 (englisch).
- ↑ Marco Marra u. a.: The Genome Sequence of the SARS-Associated Coronavirus. In: Science. Band 300, Nr. 5624, 30. Mai 2003, S. 1399–1404, doi:10.1126/science.1085953 (englisch).
- ↑ Xing-Yi Ge et al.: Isolation and characterization of a bat SARS-like coronavirus that uses the ACE2 receptor. In: Nature, Band 503, 2013, S. 535–538; doi:10.1038/nature12711
- ↑ Ben Hu, Lei-Ping Zeng, Xing-Lou Yang et al.: Discovery of a rich gene pool of bat SARS-related coronaviruses provides new insights into the origin of SARS coronavirus. In: PLoS Pathogens. 13(11): e1006698, doi:10.1371/journal.ppat.1006698
Bat cave solves mystery of deadly SARS virus — and suggests new outbreak could occur. nature.com, 1. Dezember 2017 - ↑ Maren Oldörp: SARS Übertragung, Diagnostik und Therapie.
- ↑ Pogrebnyak et al.: Severe acute respiratory syndrome (SARS) S protein production in plants: Development of recombinant vaccine. In: Proceedings of the National Academy of Sciences, 102 (25), S. 9062.
- ↑ L. Xie, Y. Liu, B. Fan et al.: Dynamic changes of serum SARS-Coronavirus IgG, pulmonary function and radiography in patients recovering from SARS after hospital discharge. In: Respir Res, 6, 5, 2005; doi:10.1186/1465-9921-6-5
- ↑ P. Zhang, J. Li, H. Liu et al.: Long-term bone and lung consequences associated with hospital-acquired severe acute respiratory syndrome: a 15-year follow-up from a prospective cohort study. In: Bone Res, 8, 8, 2020; doi:10.1038/s41413-020-0084-5
- ↑ WHO issues a global alert about cases of atypical pneumonia. WHO, 12. März 2003, abgerufen am 22. Dezember 2012 (englisch).
Auf dieser Seite verwendete Medien
Virions of family Coronaviridae (electron microscope image). Human coronavirus 229E as stated by the CDC, not SARS