Nudiviridae
| Nudiviridae | ||||||||
|---|---|---|---|---|---|---|---|---|
 Nudiviridae | ||||||||
| Systematik | ||||||||
| ||||||||
| Taxonomische Merkmale | ||||||||
| ||||||||
| Wissenschaftlicher Name | ||||||||
| Nudiviridae | ||||||||
| Links | ||||||||
|
Die Nudiviridae sind eine Familie von Viren (Nudiviren). Als natürliche Wirte dienen Pancrustacea (Insekten und marine Krebstiere). Mit Stand Juni 2021 gibt es in dieser Familie vier vom International Committee on Taxonomy of Viruses (ICTV) bestätigte Gattungen (Alphanudivirus bis Deltanudivirus). Die Infektion mit diesen Viren bedeutet für ihre Wirte chronische Erkrankung bei Adulten und Tod bei Larven.[2][1]
Die Familie ist Mitglied der 2021 neu geschaffenen Ordnung Lefavirales in der ebenfalls neuen Klasse Naldaviricetes. Diese Taxonomie löst provisorische Bezeichnungen ab wie beispielsweise „Baculo-like viruses“.[3]
Die Vorsilbe ‚nudi‘ ist abgeleitet aus dem lateinischen Wort für ‚nackt‘ und deutet an, dass die Nudiviridae nicht wie die mit ihnen verwandten Baculoviridae ihre Virionen (Viruspartikel) in Okklusionskörperchen gebündelt einschließen (englisch non-occluded).[2]
Aufbau


Die Virionen (Virusteilchen) der Nudiviridae besitzen eine stäbchenförmige Geometrie und sind behüllt. Im Gegensatz zu den ansonsten ähnlichen Baculoviridae (mit ebenfalls stäbchenförmigen Virionen) sind diese aber nicht in Okklusionskörperchen verpackt, sondern ‚nackt‘.[2][4]
Das Genom der Nudiviridae besteht aus ringförmiger (zirkulärer) Doppelstrang-DNA (dsDNA).[2]
Vermehrungszyklus
Die Replikation der Viren geschieht im Zellkern (nukleär). Die Methode der Transkription ist DNA-gestützt. Das Virus verlässt die Wirtszelle via Zusammenbruch der Kernhülle oder Export durch die Kernporen. Die Übertragungswege sind elterlich (von Müttern auf ihre Nachkommen) und sexuell.[2]
- Intranukleäre Assemblierung und Virionen-Transport am Beispiel von Oryctes rhinoceros nudivirus (OrNV)
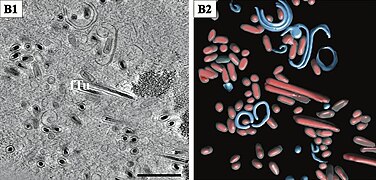
- Lange gefüllte Tubuli (fTu) und von Membranen umhüllte Virionencluster. Balken 500 nm
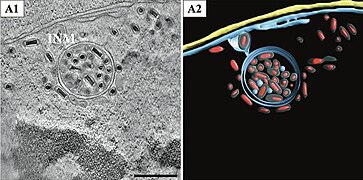
- In einem Vesikel mit Doppelmembran eingeschlossene Virionen. Balken 500 nm.
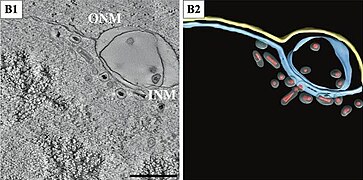
- Virionen innerhalb eines erweiterten Lumens der Kernhülle (NE). Balken 500 nm.
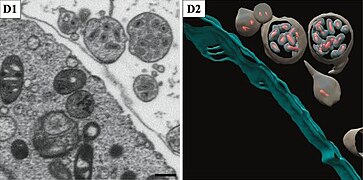
- Virionen, die in Multimembranvesikel (MMVs) im Zytoplasma eingekapselt sind. Balken 500 nm.
- Vesikel, die vollständig umhüllte Virionen enthalten. Balken 500 nm.

Wirte
Die Wirte der Gattung Alphanudivirus sind Taufliegen, Käfer (Coleoptera) und Grillen (Grylloidea), die der Gattung Betanudivirus sind Schmetterlinge (Lepidoptera).[2] Die Wirte der Gattung Gammanudivirus sind Zehnfußkrebse, die von Deltanudivirus Kohlschnaken (s. u.).
Histone
Unter den Nudiviridae gibt es offenbar Vertreter, die Histone oder Histon-Homologe kodieren. 2022 wurde die Rolle von Histonen im Replikationszyklus einer Reihe von Viren umfassend untersucht.[5]
Systematik
Innere Systematik
Mit Stand Juni 2021 sind vom ICTV folgende Spezies bestätigt:[1]
- Familie: Nudiviridae
- Gattung: Alphanudivirus[6]
- Spezies: Drosophila melanogaster nudivirus A (DiNV-A, früher Drosophila innubia nudivirus) – Wirte: Taufliegen[7][8]
- Spezies: Drosophila melanogaster nudivirus B (DiNV-B) – Wirte: Taufliegen
- Spezies: Drosophila melanogaster nudivirus C (DiNV-C) – Wirte: Taufliegen
- Spezies: Drosophila melanogaster nudivirus D (DiNV-D) – Wirte: Taufliegen
- Spezies: Gryllus bimaculatus nudivirus (GbNV) – Wirte: Grillen
- Spezies: Oryctes rhinoceros nudivirus (OrNV, Typusspezies) – Wirte: Riesenkäfer (Gattung Oryctes)
- Gattung: Betanudivirus[9]
- Spezies: Heliothis zea nudivirus (HzNV, Typusspezies) – Wirte: Schmetterlinge (Baumwollkapselbohrer)
- Spezies: „Heliothis zea nudivirus 2“ (HzNV-2, Vorschlag)[10]
- Gattung: Gammanudivirus
- Spezies: Homarus gammarus nudivirus – Wirte: Hummer
- Spezies: Penaeus monodon nudivirus (PmNV) – Wirte: Giant Tiger Prawn (Penaeus monodon)[10]
- Gattung: Deltanudivirus
- Spezies: Tiplua oleracea nudivirus (ToNV) – Wirte: Kohlschnaken[7]
Offenbar gibt es auch Insekten mit einem Genom, das integrierte (‚endogene‘) Nudiviren enthält: So etwa die braune Spornzikade Nilaparvata lugens (Nlu, englisch brown planthopper, BPH) mit dem Nilaparvata lugens endogenous nudivirus (NlENV).[11][12] Nach Bézier et al. ist NlENV in oder bei der Gattung Alphanudivirus zu verorten.[7]
Äußere Systematik
Die Nudiviridae bilden offenbar mit den Nimaviridae, Hytrosaviridae, Baculoviridae (allesamt aus der 2021 neu geschaffenen Klasse Naldaviricetes) und der Gattung Bracovirus (der vermutet polyphyletischen) Polydnaviridae[13] eine noch unbenannte Verwandtschaftsgruppe. Ein Konsenskladogramm der Autoren Koonin et al. (2015 und 2019),[14][15] Bézier et al. (2014),[7] Yang et al. (2014),[10] sowie Kawato et al. (2018)[16][10] könnte etwa wie folgt aussehen:
| Naldaviricetes |
| ||||||||||||||||||||||||||||||||||||||||||
Im 1. Halbjahr 2021 hat dea ICTV diese Gruppe als Klasse Naldaviricetes – (noch) ohne Bracovirus – offiziell anerkannt.[1] Nach Bézier et al. (2014) geht die Gattung Bracovirus direkt aus den Nudiviridae hervor und ist eine Schwesterklade der (inzwischen vom ICTV benannten) Gattung Deltanudivirus (mit ToNV).[7]
Einzelnachweise
- ↑ a b c d e ICTV: ICTV Master Species List 2020.v1, New MSL including all taxa updates since the 2019 release, March 2021 (MSL #36)
- ↑ a b c d e f Viral Zone: Nudiviridae. ExPASy, abgerufen am 30. Juli 2019.
- ↑ SIB: Double Strand DNA Viruses, auf: ViralZone.
- ↑ Viral Zone: Baculoviridae. ExPASy, abgerufen am 31. Juli 2019.
- ↑ Paul B. Talbert, Karim-Jean Armache, Steven Henikoff: Viral histones: pickpocket’s prize or primordial progenitor? In: BMC Epigenetics & Chromatin, Band 15, Nr. 21, 28. Mai 2022; doi:10.1186/s13072-022-00454-7, PMID 35624484, PMC 9145170 (freier Volltext).
- ↑ SIB: Alphanudivirus, auf: ViralZone
- ↑ a b c d e Annie Bézier, Julien Thézé, Frederick Gavory, Julien Gaillard, Julie Poulain, Jean-Michel Drezen, Elisabeth A. Herniou; G. McFadden (Hrsg.): The Genome of the Nucleopolyhedrosis-Causing Virus from Tipula oleracea Sheds New Light on the Nudiviridae Family, in: Journal of Virology 2014, doi:10.1128/JVI.02884-14, PMID 25540386
- ↑ K. A. Dyer, J. Jaenike: Evolutionarily stable infection by a male-killing endosymbiont in Drosophila innubila: molecular evidence from the host and parasite genomes. In: Genetics. 168. Jahrgang, Nr. 3, November 2004, S. 1443–1455, doi:10.1534/genetics.104.027854, PMID 15579697, PMC 1448788 (freier Volltext).
- ↑ SIB: Betanudivirus, auf: ViralZone
- ↑ a b c d Yi-Ting Yang, Der-Yen Lee, Yongjie Wang, Jer-Ming Hu, WH. Li, JH. Leu, GD. Chang, HM. Ke, ST. Kang, SS. Lin, HH. Kou, CF. Lo: The genome and occlusion bodies of marine Penaeus monodon nudivirus (PmNV, also known as MBV and PemoNPV) suggest that it should be assigned to a new nudivirus genus that is distinct from the terrestrial nudiviruses, in: BMC genomics, 15(1):628, Juli 2014, doi:10.1186/1471-2164-15-628, PMID 25063321
- ↑ Eddy Dijkstra, Jose M. Rubio, Rory J. Post: Resolving relationships over a wide taxonomic range in Delphacidae (Homoptera) using the COI gene, in: Phylogenomics, 9. Februar 2003, doi:10.1046/j.1365-3113.2003.00203.x
- ↑ Ruo-Lin Cheng, Yu Xi, Yi-Han Lou, Zhuo Wang, Ji-Yu Xu, Hai-Jun Xu, Chuan-Xi Zhang; A. Simon (Hrsg.): Brown Planthopper Nudivirus DNA Integrated in Its Host Genome, in: Journal of Virology 2014, doi:10.1128/JVI.03166-13
- ↑ Dupuy C, Huguet E, Drezen JM: Unfolding the evolutionary story of polydnaviruses. In: Virus Res. 117. Jahrgang, Nr. 1, 2006, S. 81–89, doi:10.1016/j.virusres.2006.01.001, PMID 16460826.
- ↑ Eugene V. Koonin, Natalya Yutin: Evolution of the Large Nucleocytoplasmatic DNA Viruses of Eukaryotes and Convergent Origins of Viral Gigantism, in: Advances in Virus Research, Band 103, AP 21. Januar 2019, doi:10.1016/bs.aivir.2018.09.002, S. 167–202
- ↑ Eugene V. Koonin, Valerian V. Dolja, Mart Krupovic: Origins and evolution of viruses of eukaryotes: The ultimate modularity, in: Virology Mai 2015; 479–480. 2–25. PMC 5898234 (freier Volltext), PMID 25771806.
- ↑ Satoshi Kawato, Aiko Shitara, Yuanyuan Wang, Reiko Nozaki, Hidehiro Kondo, Ikuo Hirono; Joanna L. Shisler (Hrsg.): Crustacean Genome Exploration Reveals the Evolutionary Origin of White Spot Syndrome Virus, in: Journal of Virology 2018, doi:10.1128/JVI.01144-18, PMID 30404800
Auf dieser Seite verwendete Medien
Autor/Urheber: Sailakshmi Velamoor, Allan Mitchell, Bruno M. Humbel, WonMo Kim, Charlotte Pushparajan, Gabriel Visnovsky, Laura N. Burga, Mihnea Bostina; Anne Moscona (Editor), Lizenz: CC BY-SA 4.0
Vorgeschlagenes Modell für die Assemblierung und das Trafficking der Nudiviridae. (Schritt 1) Membranmaterial wird an den Replikationsstellen in Form kleiner Vesikel und Membranfragmente akkumuliert. Zytoplasmatische Einstülpungen (CI) ermöglichen die Existenz der Membran tief im Zellkern. (Schritt 2) Virion-Assemblierungsstellen treten neben dem virogenen Stroma (VS) auf und werden mit Tu und fTu besiedelt, aus denen die neuen Virionen herausknospen. Die innere Kernmembran (INM) bildet fingerartige Strukturen (Schritt 3), die in den Kern hineinwachsen (Schritt 4) und schließlich die vollständig gebildeten Virionen aufnehmen (Schritt 5). (Schritt 6) Mit Virionen gefüllte Vesikel werden in das NE-Lumen transportiert. Durch Knospung an der äußeren Kernmembran (ONM) (Schritt 7) werden Multimembran-Vesikel im Zytoplasma freigesetzt (Schritt 8). (Schritt 9) Innerhalb des Zytoplasmas können Virionen in multivesikuläre Körper eingekapselt werden, die in der Art von Exosomen in den Raum außeerhalb der Wirtszelle freigesetzt werden. (Schritt 10) Eine Membranfusion in diesem Kompartiment kann gelegentlich Virionen im Zytoplasma freisetzen. (Schritt 11) Eine Fusion an der Plasmamembran kann viriongefüllte Vesikel in den Raum außerhalb der Wirtszelle freisetzen.
Autor/Urheber: Sailakshmi Velamoor, Allan Mitchell, Bruno M. Humbel, WonMo Kim, Charlotte Pushparajan, Gabriel Visnovsky, Laura N. Burga, Mihnea Bostina; Anne Moscona (Editor), Lizenz: CC BY-SA 4.0
Oryctes rhinoceros nudivirus (OrNV) Virion trafficking inside the host cell. (B1) A 11-nm slice through the tomographic reconstruction indicated in inset shows virions inside an expanded NE lumen. Scale bar, 500 nm. (B2) Segmentation of B1 highlights vesicle wrapped virions inside the NE lumen.
Autor/Urheber: Sailakshmi Velamoor, Allan Mitchell, Bruno M. Humbel, WonMo Kim, Charlotte Pushparajan, Gabriel Visnovsky, Laura N. Burga, Mihnea Bostina; Anne Moscona (Editor), Lizenz: CC BY-SA 4.0
Oryctes rhinoceros nudivirus (OrNV) Virion trafficking inside the host cell. (C1) A 11-nm slice through the tomographic reconstruction of the area indicated in inset displaying virions encapsulated into multimembrane vesicles (MMVs) in the cytoplasm. Scale bar, 500 nm. (C2) Segmentation showing virions inside complex MMVs, and vesicle-free virions close to the cellular membrane. (D) An infected cell showing vesicles being expelled into the extracellular space. Scale bar, 1 μm.
Autor/Urheber: Sailakshmi Velamoor, Allan Mitchell, Bruno M. Humbel, WonMo Kim, Charlotte Pushparajan, Gabriel Visnovsky, Laura N. Burga, Mihnea Bostina; Anne Moscona (Editor), Lizenz: CC BY-SA 4.0
Intranuclear assembly of Oryctes rhinoceros nudivirus (OrNV) virions.(B1) A 11-nm-thick section showing long filled tubules (fTu) and virion clusters being enveloped in membranes. Scale bar, 500 nm. (B2) Segmentation of the tomogram displaying viral capsids (red) inside filled tubules (gray) and membrane structures (blue).
Autor/Urheber: Sailakshmi Velamoor, Allan Mitchell, Bruno M. Humbel, WonMo Kim, Charlotte Pushparajan, Gabriel Visnovsky, Laura N. Burga, Mihnea Bostina; Anne Moscona (Editor), Lizenz: CC BY-SA 4.0
Oryctes rhinoceros nudivirus (OrNV) Virion trafficking inside the host cell. (D1) A slice through the tomographic reconstruction of the area indicated in inset showing vesicles containing fully enveloped virions. Scale bar, 500 nm. (D2) Segmentation showing virions within vesicles.
Autor/Urheber: ViralZone, SIB Swiss Institute of Bioinformatics: Nudiviridae, Lizenz: CC BY 4.0
Schemazeichnung eines Virusteilchens der Familie Nudiviridae
Autor/Urheber: Sailakshmi Velamoor, Allan Mitchell, Bruno M. Humbel, WonMo Kim, Charlotte Pushparajan, Gabriel Visnovsky, Laura N. Burga, Mihnea Bostina; Anne Moscona (Editor), Lizenz: CC BY-SA 4.0
Cryo-EM of Oryctes rhinoceros nudivirus (OrNV) shed into the extracellular space showing the capsid surrounded by a thick envelope
Autor/Urheber: Sailakshmi Velamoor, Allan Mitchell, Bruno M. Humbel, WonMo Kim, Charlotte Pushparajan, Gabriel Visnovsky, Laura N. Burga, Mihnea Bostina; Anne Moscona (Editor), Lizenz: CC BY-SA 4.0
Capsid segmentation of Oryctes rhinoceros nudivirus (OrNV)
Autor/Urheber: Sailakshmi Velamoor, Allan Mitchell, Bruno M. Humbel, WonMo Kim, Charlotte Pushparajan, Gabriel Visnovsky, Laura N. Burga, Mihnea Bostina; Anne Moscona (Editor), Lizenz: CC BY-SA 4.0
Oryctes rhinoceros nudivirus (OrNV) Virion trafficking inside the host cell. (A1) A 11-nm slice through the tomographic reconstruction indicated in inset showing virions enclosed in a double membrane vesicle. Scale bar, 500 nm. (A2) Segmentation of the tomogram. (B) Micrograph displaying expanded NE. Scale bar, 1 μm.