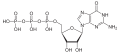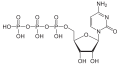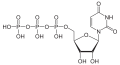Nukleotide
Als Nukleotide, auch Nucleotide (abgekürzt nt), werden die Bausteine von Nukleinsäuren sowohl in Strängen der Ribonukleinsäure (RNA bzw. deutsch RNS) wie auch der Desoxyribonukleinsäure (DNA bzw. deutsch DNS) bezeichnet. Ein Nukleotid setzt sich aus einem Basen-, einem Zucker- und einem Phosphatanteil zusammen.
Während Nukleoside nur aus dem Basen- und dem Zuckeranteil bestehen, enthalten Nukleotide zusätzlich Phosphatgruppen. Unterschiede zwischen einzelnen Nukleotidmolekülen können daher jeweils in der Nukleobase, dem Monosaccharid und dem Phosphatrest bestehen.

Ein Nukleosid ist die Verbindung einer Nukleinbase (Base) mit einem Einfachzucker, einer Pentose. Deren 2'-Rest (R) ist im Falle der Ribose eine Hydroxygruppe (OH-), im Falle der Desoxyribose hingegen Wasserstoff (H-).
Bei einem Nukleotid ist die 5'-OH-Gruppe der Pentose eines Nukleosids mit einem Phosphatrest verestert. Ein Nukleosidtriphosphat (NTP) weist drei Phosphatgruppen auf, die untereinander Säureanhydridbindungen ausbilden. Mit Adenin als Base und Ribose als Saccharid liegt das Adenosintriphosphat (ATP) vor.
Bei artifiziellen Didesoxynukleotiden ist auch die 3'-OH-Gruppe durch ein H-Atom ersetzt.
Nukleotide treten nicht nur als Monophosphate (NMP) verknüpft in den informationstragenden Makromolekülen von Nukleinsäuren auf. Sie tragen auch weitere Funktionen für die Regulation von Lebensvorgängen in Zellen. So spielen Triphosphate (NTP) wie beispielsweise Adenosintriphosphat (ATP) eine zentrale Rolle beim Energietransfer zwischen Stoffwechselwegen, als Cofaktor für die Aktivität von Enzymen, für den Transport durch Motorproteine oder die Kontraktion von Muskelzellen. Guanosintriphosphat (GTP) bindende (G-)Proteine übermitteln Signale von Membranrezeptoren, das cyclische Adenosinmonophosphat (cAMP) ist ein wichtiger intrazellulärer Botenstoff.
Aufbau eines Nukleotids
Ein Nukleotid ist aus drei Bestandteilen aufgebaut:
- Base – einer der fünf Nukleobasen, nämlich Adenin (A), Guanin (G), Cytosin (C), Thymin (T) oder Uracil (U);
- Zucker – einem Monosaccharid (Einfachzucker) mit 5 C-Atomen, der als Furanosering vorliegenden Pentose, nämlich Ribose (D-Ribofuranose) oder Desoxyribose (2-Desoxy-D-ribofuranose);
- Phosphat – einem Rest mit mindestens einer Phosphatgruppe.

Hierbei wird die Base mit dem Zucker zumeist über eine N-glykosidische Bindung verknüpft, der Zucker mit dem Phosphat über eine Esterbindung; ist mehr als eine Phosphatgruppe angehängt, so sind diese untereinander über Phosphorsäureanhydridbindungen verknüpft. Daneben kommen auch Nukleotide natürlich vor, in denen Zucker und Base über eine C-glykosidische Bindung verknüpft sind, beispielsweise das Pseudouridin (Ψ) in Transfer-RNA.
Die DNA besteht aus den vier Nukleinbasen A, G, C, T. Anstelle des DNA-Bausteins Thymin wird in der RNA das Uracil eingesetzt. Somit enthält die RNA die vier Basen A, G, C, U. Die einzelnen Nukleotide unterscheiden sich jeweils durch die Base und durch den Zucker, die namensgebende Pentose, die bei der DNA Desoxyribose und bei der RNA Ribose ist.
Verknüpfung von Nukleotiden zu Nukleinsäuren
Das Makromolekül einer DNA oder einer RNA ist jeweils aus vier verschiedenen Sorten von Nukleotiden zusammengesetzt, die durch kovalente Bindungen zum Strang des polymeren Biomoleküls verknüpft werden, einem Polynukleotid. Die hierbei ablaufende Reaktion ist eine Kondensationsreaktion. Von den monomeren Nukleosidtriphosphaten wird dabei ein Pyrophosphatrest abgespalten, sodass die Monosaccharide der Nukleotide über je eine Phosphatgruppe aneinander gekoppelt werden, die das C5'-Atom der nächsten mit dem C3'-Atom der vorangehenden Pentose verbindet.

Mit diesem Pentose-Phosphat-Rückgrat wird der Einzelstrang einer Nukleinsäure aufgebaut, also mit Desoxyribose-Phosphat bei einer DNA. Bei einem DNA-Doppelstrang liegen die Basen der Nukleotide des einen DNA-Einzelstrangs den Basen der Nukleotide des anderen Einzelstrangs gegenüber; deren Phosphat-Desoxyribose-Rückgrat zeigt somit nach außen.
Typischerweise bilden dabei jeweils eine (kleinere) Pyrimidinbase (T, C) und eine (größere) Purinbase (A, G) ein Paar. Als komplementär werden die Basenpaare aus T und A sowie aus C und G bezeichnet: Gegenüber einem Nukleotid, das Cytosin als Base beinhaltet, liegt in der Regel ein Nukleotid mit Guanin als Base (und umgekehrt); das Gleiche gilt für das Basenpaar aus Adenin und Thymin. Die einander gegenüberliegenden Basen der Nukleotide zweier Stränge sind in der DNA-Doppelhelix über Wasserstoffbrückenbindungen miteinander verbunden. Zwischen den Basen G und C bilden sich drei, zwischen A und T nur zwei.
Dieser Basenpaarungsmechanismus erlaubt nicht nur die Formierung von DNA-Helices. Indem den Basen eines Einzelstrangs je die komplementäre Base zugeordnet wird, wird es auch möglich, einen komplementären Einzelstrang neu aufzubauen. Dies geschieht beispielsweise bei der Replikation mithilfe einer DNA-Polymerase.
RNA-Moleküle sind ebenfalls aus Nukleotiden aufgebaut, mit dem Unterschied, dass hier Ribose statt Desoxyribose als Monosaccharid verwendet wird, und dass Uracil anstelle von Thymin als Base auftritt. Die geringen Unterschiede im Gerüst von einzelsträngigen RNA- und DNA-Molekülen hindern sie nicht daran, ebenso zwischen komplementären Basen Wasserstoffbrücken auszubilden. Auch sind komplementäre Basenpaarungen innerhalb desselben Strangmoleküls möglich. Beispielsweise können sich darüber bestimmte Abschnitte eines RNA-Moleküls zu Haarnadelstrukturen aneinanderlegen und falten. Auch mehrfache Schleifenbildungen sind möglich, für die Ausbildung der Kleeblattstruktur von tRNA-Molekülen sogar typisch. Obgleich RNA also auch doppelsträngig auftreten kann, auch als Helix, bestehen die meisten der biologisch aktiven RNA-Moleküle aus einem einzelnen Strang.
Drei miteinander verbundene Nukleotide stellen dabei die kleinste Informationseinheit dar, die in DNA oder in RNA zur Codierung der genetischen Information zur Verfügung steht. Man nennt diese Informationseinheit ein Codon.
Nukleotide als Mono-, Di- und Triphosphate
Nukleotide bestehen aus einem Nukleosid und einem Rest aus ein, zwei oder drei Phosphatgruppen.
- Nukleobase + Pentose = Nukleosid
- Nukleobase + Pentose + 1 Phosphatgruppe = Nukleotid (Nukleosidmonophosphat, NMP)
- Nukleobase + Pentose + 2 Phosphatgruppen = Nukleotid (Nukleosiddiphosphat, NDP)
- Nukleobase + Pentose + 3 Phosphatgruppen = Nukleotid (Nukleosidtriphosphat, NTP)
Ribose als Zucker
Desoxyribose als Zucker
| Nukleobase | Nukleosid | Nukleotid | Strukturformel | ||
|---|---|---|---|---|---|
| Adenin | A | Desoxyadenosin | Desoxyadenosinmonophosphat | dAMP |  |
| Desoxyadenosindiphosphat | dADP |  | |||
| Desoxyadenosintriphosphat | dATP |  | |||
| Guanin | G | Desoxyguanosin | Desoxyguanosinmonophosphat | dGMP |  |
| Desoxyguanosindiphosphat | dGDP |  | |||
| Desoxyguanosintriphosphat | dGTP | 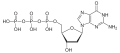 | |||
| Cytosin | C | Desoxycytidin | Desoxycytidinmonophosphat | dCMP |  |
| Desoxycytidindiphosphat | dCDP | 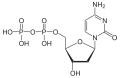 | |||
| Desoxycytidintriphosphat | dCTP | 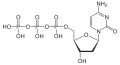 | |||
| Thymin | T | Desoxythymidin | Desoxythymidinmonophosphat | dTMP |  |
| Desoxythymidindiphosphat | dTDP |  | |||
| Desoxythymidintriphosphat | dTTP |  | |||
Didesoxyribose als Zucker
Artifizielle Didesoxyribonukleosidtriphosphate (ddNTPs) finden beispielsweise Verwendung bei der DNA-Sequenzierung nach Sanger. Didesoxynukleosid-Analoga wurden in den 1980er Jahren[1] als Infektions-Inhibitoren bereitgestellt[2] zur Therapie bei HIV-Infektionen.
Notation von Nukleotiden
Für die Notation der Basen von Nukleotiden in Nukleinsäuresequenzen werden Buchstabensymbole verwendet. Um auch Mehrdeutigkeiten (engl.: ambiguity) unvollständig spezifizierter Nukleobasen berücksichtigen zu können, wurde vom Nomenklaturkomitee der International Union of Biochemistry and Molecular Biology der Ambiguity Code vorgeschlagen:[3]
| Symbol | Bedeutung | Herleitung |
|---|---|---|
| G | G | Guanin |
| A | A | Adenin |
| C | C | Cytosin |
| T | T | Thymin |
| U | U | Uracil |
| R | G oder A | Purine |
| Y | C oder T | Pyrimidine |
| W | A oder T | schwach (engl. weak) mit 2 Wasserstoffbrücken gepaart |
| S | G oder C | stark (engl. strong) mit 3 Wasserstoffbrücken gepaart |
| M | A oder C | Aminogruppe |
| K | G oder T | Ketogruppe |
| H | A, C, oder T (U) | nicht G, im Alphabet folgt H auf G |
| B | G, C, oder T (U) | nicht A, im Alphabet folgt B auf A |
| V | G, A, oder C | nicht T (U), im Alphabet folgt V auf U |
| D | G, A, oder T (U) | nicht C, im Alphabet folgt D auf C |
| N | G, A, C oder T (U) | irgendeine (engl. any) der Basen |
Man beachte den Unterschied zwischen den obigen generischen Symbolen W, S,… und den so genannten Wobble-Basen. Modifikationen der obigen Symbole und auch zusätzliche Symbole gibt es für Nicht-Standard-Basen und modifizierte Basen, etwa den griechischen Buchstaben Psi, Ψ für Pseudouridin (eine Wobble-Base).
Etliche der obigen Symbole finden allerdings auch alternativ für synthetische Basen Verwendung, siehe DNA §Synthetische Basen. Eine Auswahl:
- P – 2-Amino-imidazo[1,2-a]-1,3,5-triazin-4(8H)-on und Z – 6-Amino-5-nitro-2(1H)-pyridon[4][5][6]
- X – NaM, Y – 5SICS und Y' – TPT3![7][8] Weitere Basen aus dieser Serie: FEMO und MMO2[9]
- P – 5-Aza-7-deazaguanin, B – Isoguanin, rS – Isocytosin, dS – 1-Methylcytosin und Z – 6-Amino-5-nitropyridin-2-on, siehe Hachimoji-DNA[10][11]
- xA, xT, xC, xG (analog mit Präfix xx, y und yy), siehe xDNA[12]
Funktionen von Nukleotiden
Neben der Funktion der Nukleotide als Grundbausteine im Polymer von Nukleinsäuren der DNA und RNA,[13] erfüllen Nukleotide weitere Funktionen als einzelne Moleküle, monomer, und spielen so bei der Regulation von Lebensvorgängen eine wichtige Rolle. Beispiele hierfür finden sich zahlreich im Energietransfer zwischen Stoffwechselwegen der Zelle. Monomere Nukleotide treten auch als Cofaktoren von Enzymen auf, so etwa im Coenzym A.[14]
Nukleotide lassen sich energetisch nach der Anzahl der Phosphatgruppen unterscheiden als Monophosphate (NMP), Diphosphate (NDP) oder Trisphosphate (NTP) der jeweiligen Nukleoside. Beispielsweise entsteht aus Adenosintriphosphat (ATP) durch Abspaltung eines Phosphatrestes Adenosindiphosphat (ADP), oder durch Abspaltung von Pyrophosphat das energetisch minderwertigere Adenosinmonophosphat (AMP). Das cyclische AMP (cAMP) spielt daneben eine bedeutende Rolle bei der Signaltransduktion in einer Zelle, als sogenannter sekundärer Botenstoff (englisch second messenger).
Siehe auch
- Dinukleotid
- Oligonukleotid
- Polynukleotid
- cyclisches Guanosinmonophosphat (cGMP)
- Xanthosinmonophosphat (XMP)
- Flavinmononukleotid (FMN)
Literatur
- D. Voet, J. Voet, C. Pratt: Lehrbuch der Biochemie. 2. Auflage. Wiley-VCH, Weinheim 2010, ISBN 978-3-527-32667-9, S. 45ff, 600ff.
- Bruce Alberts u. a.: Lehrbuch der Molekularen Zellbiologie. 4. Auflage. Wiley, 2012, ISBN 978-3-527-32824-6, S. 57–62, 80ff.
Einzelnachweise
- ↑ E. Piechowak: Wo kann man das HIV-Virus treffen? Interventionsstationen antiviraler Therapie. In: Fortschritte der Medizin. Band 105, 1987, Nr. 10, S. 34–36.
- ↑ Gundolf Keil: Umgang mit AIDS-Kranken als Herausforderung an eine humane Gesellschaft. „Statement“ zum Akquirierten Immun-Defizienz-Syndrom aus fachhistorischer Perspektive. In: Johannes Gründel (Hrsg.): AIDS. Herausforderung an Gesellschaft und Moral. 2. Auflage. Düsseldorf 1988 (= Schriften der Katholischen Akademie in Bayern. Band 125), S. 31–41, hier: S. 41.
- ↑ Nomenclature Committee of the International Union of Biochemistry (NC-IUB): Nomenclature for Incompletely Specified Bases in Nucleic Acid Sequences. 1984, abgerufen am 16. November 2019.
- ↑ A. M. Sismour, S. Lutz, J. H. Park, M. J. Lutz, P. L. Boyer, S. H. Hughes, S. A. Benner: PCR amplification of DNA containing non-standard base pairs by variants of reverse transcriptase from Human Immunodeficiency Virus-1., in: Nucleic Acids Research. Band 32, Nummer 2, 2004, S. 728–735, doi:10.1093/nar/gkh241, PMID 14757837, PMC 373358 (freier Volltext).
- ↑ Z. Yang, D. Hutter, P. Sheng, A. M. Sismour und S. A. Benner: Artificially expanded genetic information system: a new base pair with an alternative hydrogen bonding pattern. (2006) Nucleic Acids Res. 34, S. 6095–6101. PMC 1635279 (freier Volltext)
- ↑ Z. Yang, A. M. Sismour, P. Sheng, N. L. Puskar, S. A. Benner: Enzymatic incorporation of a third nucleobase pair., in: Nucleic Acids Research. Band 35, Nummer 13, 2007, S. 4238–4249,
doi:10.1093/nar/gkm395, PMID 17576683, PMC 1934989 (freier Volltext). - ↑ Yorke Zhang, Brian M. Lamb, Aaron W. Feldman, Anne Xiaozhou Zhou, Thomas Lavergne, Lingjun Li, Floyd E. Romesberg: A semisynthetic organism engineered for the stable expansion of the genetic alphabet, in: PNAS 114 (6), 7. Februar 2017, S. 1317–1322; Erstveröffentlichung 23. Januar 2017, doi:10.1073/pnas.1616443114, Hrsg.: Clyde A. Hutchison III, The J. Craig Venter Institute
- ↑ scinexx: Forscher züchten „Frankenstein“-Mikrobe, vom 24. Januar 2017.
- ↑ Indu Negi, Preetleen Kathuria, Purshotam Sharma, Stacey D. Wetmore: How do hydrophobic nucleobases differ from natural DNA nucleobases? Comparison of structural features and duplex properties from QM calculations and MD simulations, in: Phys. Chem. Chem. Phys. 2017, 19, S. 16305–16374, doi:10.1039/C7CP02576A
- ↑ Shuichi Hoshika, Nicole A. Leal, Myong-Jung Kim, Myong-Sang Kim, Nilesh B. Karalkar, Hyo-Joong Kim, Alison M. Bates, Norman E. Watkins Jr., Holly A. SantaLucia, Adam J. Meyer, Saurja DasGupta, Joseph A. Piccirilli, Andrew D. Ellington, John SantaLucia Jr., Millie M. Georgiadis, Steven A. Benner: Hachimoji DNA and RNA: A genetic system with eight building blocks. Science 363 (6429), 22. Februar 2019, S. 884–887, doi:10.1126/science.aat0971.
- ↑ Daniela Albat: DNA mit acht Buchstaben, auf: scinexx und Erweiterter Code des Lebens, auf: wissenschaft.de (bdw online), beide vom 22. Februar 2019.
- ↑ S. R. Lynch, H. Liu, J. Gao, E. T. Kool: Toward a designed, functioning genetic system with expanded-size base pairs: solution structure of the eight-base xDNA double helix., in: Journal of the American Chemical Society. Band 128, Nr. 45, November 2006, S. 14704–14711, doi:10.1021/ja065606n. PMID 17090058. PMC 2519095 (freier Volltext).
- ↑ Nukleotide – Biologie. Abgerufen am 13. Februar 2020.
- ↑ Tim Bugg: Introduction to enzyme and coenzyme chemistry. 3rd Auflage. Wiley, Chichester, West Sussex 2012, ISBN 978-1-118-34899-4.
Auf dieser Seite verwendete Medien
Autor/Urheber:
- DNA_chemical_structure.svg: Madeleine Price Ball, User:Madprime
- derivative work: Matt (talk)
Chemische Struktur der DNA mit farbigen Beschriftungen um die 4 Basen, die Phosphate und die Desoxyribose zu kennzeichnen.
Struktur von Guanosindiphosphat
Struktur von Desoxycytidindiphosphat
Struktur von Desoxycytidinmonophosphat
Struktur von Desoxyadenosindiphosphat
Struktur von Adenosindiphosphat
Struktur von Desoxythymidindiphosphat
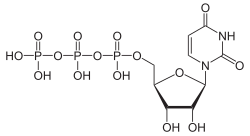
Struktur von Cytidintriphosphat
Struktur von Uridintriphosphat
Struktur von Uridindiphosphat
Struktur von Cytidinmonophosphat
Autor/Urheber: Yikrazuul, Lizenz: CC BY-SA 3.0
Allgemeiner Aufbau eines Nucleosids und eines Nucleotids
Struktur von Desoxycytidintriphosphat
Struktur von Uridinmonophosphat
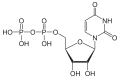
Struktur von Desoxythymidinmonophosphat
Struktur von Adenosintriphosphat (ATP), protonierte Form
Struktur von Desoxyguanosindiphosphat
Struktur von Desoxyadenosinmonophosphat
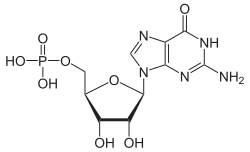
Struktur von Guanosinmonophosphat
Struktur von Guanosintriphosphat
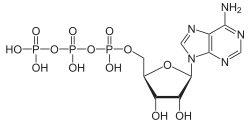
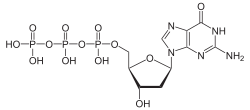
Struktur von Desoxyadenosintriphosphat
Struktur von Cytidindiphosphat
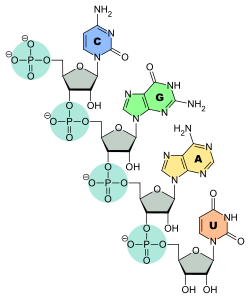
Verknüpfung der Nukleinbasen C, G, A und U mit dem Ribosephosphat-Rückgrat der RNA
Struktur von Desoxyguanosinmonophosphat
Struktur von Adenosinmonophosphat
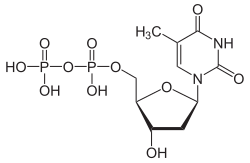
Struktur von Desoxythymidintriphosphat
Struktur von Desoxyguanosintriphosphat