Megavirus
| Mimiviridae Gruppe I Linie C | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
 Megavirus-Virion | ||||||||||||||||||
| Systematik | ||||||||||||||||||
| ||||||||||||||||||
| Taxonomische Merkmale | ||||||||||||||||||
| ||||||||||||||||||
| Wissenschaftlicher Name | ||||||||||||||||||
| Megavirus | ||||||||||||||||||
| Kurzbezeichnung | ||||||||||||||||||
| MGV | ||||||||||||||||||
| Links | ||||||||||||||||||
|
Megavirus ist eine im April 2023 vom International Committee on Taxonomy of Viruses (ICTV) aufgestellte Gattung von Riesenviren in der Familie Mimiviridae.[1] Die Gattung ersetzte die vormals in der Schwestergattung Mimivirus als „Linie C“ alias „Courdo11-Gruppe“ zusammengefassten isolierten Viren und vorgeschlagenen Virusspezies. Die Gattung umfasst seitdem die Spezies Megavirus boshanense, Megavirus powaiense und Megavirus chilense; zur letzteren Art gehört der erste gefundene Vertreter der Gattung, Megavirus chilensis (MVC, MVc oder MGVC; öfters als Megavirus chiliensis verschrieben), der 2010 von französischen Wissenschaftlern vor der Küste von Chile entdeckt wurde,[4] sowie Megavirus courdo11. Zur Art Megavirus powaiense gehört Powai lake megavirus isolate 1, zu Megavirus boshanense gehört Megavirus baoshan strain SH.[5]
MGVC ist ein Verwandter von Acanthamoeba polyphaga mimivirus (APMV, Mimivirus bradfordmassiliense)[6] und infiziert ebenfalls Acanthamöben. Wegen der nur relativ entfernten Verwandtschaft[4] hat das ICTV im April 2023 die als Megavirus identifizierten Viren, die früher als Linie C vorschlagsgemäß ebenfalls der Gattung Mimivirus zugerechnet wurden, in eine eigene Gattung Megavirus (MGV) verschoben.[4][5][1]
Innerhalb der Familie der Mimiviridae wird die Gattung Megavirus zusammen mit den Gattungen Mimivirus und Moumouvirus der „Gruppe I“ zugerechnet, die die Unterfamilie Megamimivirinae inklusive Mimivirus umfasst.[1] Das ICTV ist damit im Prinzip früheren Vorschlägen wie von Schulze et al. gefolgt,[2] und diesen den Verzug gegeben gegenüber anderen Vorschlägen wie „Mimivirinae“,[7] und „Megavirinae“.[8]
Wie die Mitglieder der Gattungen Mimivirus und Moumouvirus können auch Viren der Gattung Megavirus von Virophagen befallen werden, z. B. von „Sputnik 3“ und Zamilon.[9][10][11]
Entdeckung
Megavirus chilensis (MVC) wurde aus Meerwasser isoliert, das im April 2010 vor der Küste von Chile in der Nähe von Las Cruces von Jean-Michel Claverie und Chantal Abergel von der Universität Aix-Marseille gesammelt wurde. Wissenschaftler aus diesem Labor waren auch an der Charakterisierung des Mimivirus ApMV beteiligt. Das Virus wurde per der Co-Kultivierung mit einer Reihe von Acanthamöben-Stämmen isoliert, und zwar von den Spezies (Acanthamoeba polyphaga, A. castellanii und A. griffini). Dabei wurde ein Verfahren benutzt, das von Timothy Rowbotham für die Isolierung intrazellulärer parasitischer Bakterien entwickelt worden war.[12] Der natürliche Wirt des MGVC ist vermutlich ein phagozytisches Protozoon, das im Meer- oder Brackwasser lebt.
Morphologie
Die Virionen (Virusteilchen) des MGVC bestehen aus einem Protein-Kapsid mit einem Durchmesser von 440 nm. Sie erreichen damit fast die Ausmaße kleiner Bakterien und gehören damit zu den größten beschriebenen Viren. Die Virionen sind von einem mehrlagigen Tegument-Material von 75 bis 100 nm Schichtdicke umgeben (vgl. Mimivirus-Proteinfilamente). Das Kapsid erscheint hexagonal, allerdings ist seine ikosaedrische Symmetrie nicht perfekt, da es (mindestens ein) sogenanntes „Stargate“ enthält. Als „Stargate“ wird eine Struktur bezeichnet, die einem fünfzackigen Stern ähnelt und die eine Öffnung bildet, durch die das Kernmaterial (d. h. die Genom-DNA) des Virus in das Zytoplasma des Wirts gelangt-.[13] Das Kapsid ist von zwei Membranen umgeben, die unterschiedliche virale Proteine enthalten.
Genom
Das Genom von MGVC besteht aus einer linearen Doppelstrang-DNA mit einer Länge von 1.259.197 bp (Basenpaaren); damit war es zur Zeit seiner Entdeckung das größte bekannte Virusgenom. Es ist um 67,5 kbp größer als das Genom des Mimivirus ApMV und enthält vermutlich 1120 kodierende Regionen (i. e. kodierte Proteine), was mehr ist als bei vielen Bakterien. Der GC-Gehalt liegt bei 25 %.[14]
MGVC besitzt sieben Aminoacyl-tRNA-Synthetasen, Enzyme, die üblicherweise nur in zellulären Organismen vorkommen. Vier dieser Proteine sind auch bekannt aus Acanthamoeba polyphaga mimivirus (ApMV) und dem Acanthamoeba castellanii mamavirus (ACMV), beides Viren der Spezies Mimivirus bradfordmassiliense; nämlich diejenigen für die Aminosäuren Tyrosin, Arginin, Cystein und Methionin. MGVC enthält aber noch zusätzlich die Aminoacyl-tRNA-Synthetasen für Tryptophan, Asparagin und Isoleucin. Das Cafeteria-roenbergensis-Virus (CroV, Rheavirus sinusmexicani) enthält nur die Aminoacyl-tRNA-Synthetase für Isoleucin. Das MGVC enthält darüber hinaus das Gen für eine besondere Variante des mismatch repair Enzyms MutS, das sonst nur in den Mitochondrien von Octocorallia-Blumentiere vorkommt. Diese MutS-Version scheint nur bei Mitgliedern der Familie Mimiviridae vorzukommen.[15] MGVC enthält – ähnlich wie ApMV – Gene für den Stoffwechsel von Zuckern, Fetten und Aminosäuren.[16]
MGVC und ApMV haben 594 orthologe Gene gemeinsam. Die meisten dieser übereinstimmenden Gene befinden sich in der Mitte der viralen Genome, jeweils flankiert von artspezifischen Abschnitten im 5′- und 3′-Bereich. Bezüglich der Aminosäuresequenzen sind die durch diese orthologen Gene codierten Proteine zu etwa 50 % identisch.
Aus der Analyse des Virusgenoms von MGVC ergaben sich Hinweise darauf, dass Mimiviren und Megaviren (d. h. die Linien A und C) von einem gemeinsamen Vorfahren abstammen könnten, der durch reduktive Evolution aus Bestandteilen eines zellulären Genoms entstanden sei.[17]
Replikation

Die Replikationsstadien von MGVC ähneln denjenigen des Mimivirus ApMV. Kurz nach der Phagozytose und der Freisetzung des inneren Kapsids in das Zytoplasma der Wirtszelle beginnt die Eklipse-Phase. Man findet dabei sog. zytoplasmatische „Seeds“, die in ihrer Größe dem Megavirus-Kern (ohne die Hülle) entsprechen und sich innerhalb von 14 Stunden zu Virion-Fabriken entwickeln.[A. 1] Bis zur Lyse der Amöbenzelle und der Freisetzung der Viren dauert es beim MGVC gewöhnlich ca. 17 Stunden (im Gegensatz zu ca. 12 Stunden bei Mimivirus ApMV). Es werden dabei ca. 500 neue Viren pro infizierter Zelle produziert – im Gegensatz zu 1000 Partikeln bei ApMV.
MGVC gehört damit zum Phylum Nucleocytoviricota, einer Gruppe von großen zytoplasmatischen Viren, früher auch bekannt als Nucleocytoplasmic large DNA viruses (NCLDV). Als Klassifikationsmerkmal dieses Phylums gilt, dass zugehörigen Viren sich vollständig im Zytoplasma des Wirtes mithilfe sogenannter large virion factories (großer Virus-Fabriken) replizieren, ohne (wie sonst bei dsDNA-Viren üblich) ihr Genom mithilfe der wirtseigenen DNA-Polymerasen im Zellkern der Wirtszelle zu replizieren.
Bedeutung der Entdeckung
Zwei Eigenschaften von MGVC sind von Bedeutung: Nur 6 % größer als ApMV, so war bereits bei seiner Entdeckung anzunehmen, dass es nicht das größte Virus ist. Die Tatsache, dass drei weitere Aminoacyl-tRNA-Synthetasen in Virengenomen gefunden wurden, ließ die Vermutung aufkommen, dass diese Enzyme nicht durch einen lateralen Gentransfer von anderen Viren erworben (Virus zu Virus, setzt Doppelinfektion voraus) wurden. Die Analyse Der DNA-Sequenz zeigte dagegen Verwandtschaft mit eukaryotischen Genen an. Davon ausgehend wurde vermutet, dass das Genom dieser Viren ähnlich wie bei anderen Parasiten durch Genomreduktion aus dem Genom eines zellulären Organismus entstanden sein könnte (Wirt zu Virus, englisch host to virus, HtoV oder H2V). Weiterhin wurde vermutet, dass MGVC (bzw. die Gattung Megavirus) evolutionär sehr alt sei, und zwar eventuell älter als die heutigen Eukaryoten. Sie könnten dann gleichzeitig mit dem Zellkern entstanden sein; eine Vermutung, die durch die Ähnlichkeit des Zellkerns mit der oben erwähnten Virusfabrik unterstützt wird. Eine weitere Vermutung war, dass diese Riesenviren von einer zwischenzeitlich ausgestorbenen zellulären Domäne abstammen.[18] Diesen Ansichten wurde jedoch widersprochen und argumentiert, dass die Nucleocytoviricota (NCLDV) ihre Größe vermutlich auf mehreren Linien unabhängig durch Acquisition von Genen per horizontalem Gentransfer (H2V) erworben haben.[19] Durch die Entdeckung von Medusavirus wurde die Frage allerdings wieder neu aufgeworfen, da diese sich von anderen NCLDVs stark unterschieden, d. h. dass Gattung im Stammbaum der NCLDVs basal zu stehen schien, Das ICTV hat die Gattung Medusavirus im April 2023 dann der NCLDv-Klasse Megaviricetes ohne nähere Klassifizierung zugeordnet.[1]
Systematik



Mit der Reorganisation der Ordnung Imitervirales durch das IVTC Anfang April 2023 wurden in der Familie Mimiviridae drei Unterfamilie, darunter die Megamimivirinae eingerichtet, sowie in dieser neben der bereits bestehenden Gattung Mimivirus weitere, darunter Cotonvirus, Moumouvirus, Tupanvirus und Megavirus- Diese neuen Gattungen ersetzen auch die frühere provisorische Aufteilung der Gattung Mimivirus in Subkladen (bezeichnet als Mimivirus-Linien A, B und C). Frühere Zuordnungen von Kandidaten zur Gattung Mimivirus (wie z. B. „Mimivirus LCMiAC01“ und „02“ oder „gvSAG AB-566-O17“) sind in diesem Licht nicht mehr verbindlich, denn diese Vorschläge könnten jetzt in eine der neuen Gattungen zu klassifizieren sein.[5]
Die hier wiedergegebene Systematik basiert auf der Master Species List (MSL) #38 des International Committee on Taxonomy of Viruses (ICTV) vom 8. April 2023[1] und dem vorausgegangenen Vorschlag zur Reorganisation der Ordnung Imitervirales durch Aylward et al. (2021),[5] ergänzt um Vorschläge in doppelten Anführungszeichen nach der NCBI-Taxonomie vom 1. Mai 2023:[20]
- Unterfamilie Megamimivirinae (Vorschlag Schulz et al., 2018,[2] zu Mimiviridae Gruppe I, Mimiviren s. l., früher auch „Mimivirinae“ genannt[7])
- Gattung Megavirus (Mimiviridae: Gruppe I Linie C, MC,[21] Courdo11-Linie oder Megavirus-Linie)
- Spezies Megavirus baoshanense mit
Megavirus baoshan strain SH - Spezies Megavirus chilense[1][5] (Schreibvariante Megavirus chiliense[5]) mit
Megavirus chilensisT (Schreibvariante Megavirus chiliensisT)[22][23][24]
Megavirus lba isolate LBA111,[25][26][27]
Megavirus terra1 (kurz Terravirus 1),[28][27]
Megavirus vitis isolate vigne – isoliert aus dem Boden eines französischen Weinbergs bei Marseille,[21][29][22]
„Acanthamoeba polyphaga mimivirus“ ASM381511v1 (verschoben),
„Acanthamoeba polyphaga mimivirus“ ASM381513v1 (verschoben),
Megavirus courdo11 (kurz Courdovirus 11),[28][30][27] – vgl. auch Courdo virus CE11[31][32]
„Megavirus courdo5“ (kurz „Courdovirus 5“),[33][27]
„Megavirus courdo7“ (kurz „Courdovirus 7“),[34][30][27][23]
„Megavirus vitis transpoviron“ (mvtv)[21] - Spezies Megavirus powaiense (Powai lake megavirus, PLMV) mit
Powai lake megavirus isolate 1[30][27][35][22][36][37] - Spezies „Megavirus avenue9“[38][27]
- Spezies „Megavirus balcon“[39][27]
- Spezies „Megavirus battle43“[40][27]
- Spezies „Megavirus bus“[41][27] (veraltet: Mimivirus bus[42][43])
- Spezies „Megavirus feuillage“[44]
- Spezies: „Megavirus J3“[45][27]
- Spezies „Megavirus mammoth“[46]
- Spezies „Megavirus mont1“[47][28]
- Spezies: „Megavirus montpellier“[48](alias „Megavirus montpellier3“[27])
- Spezies: „Megavirus potager“[49]
- Spezies: „Megavirus ursino“[50][30][27]
- Spezies: „Megavirus shan“ (alias „Shan-Virus“)[51][26][27]
- Spezies: „Megavirus T1“[52][27]
- Spezies: „Megavirus T4“[53][27]
- Spezies: „Megavirus T6“[54][27]
- ?Spezies: „Bandra megavirus“ (BMV)[55][35]
- ?Spezies: „Afrovirus urmite69“[56][10]
- ohne Spezieszuweisung:
Megavirus musashi – erstmals gefunden in der Präfektur Saitama, Japan; infiziert Acanthamoeba castellanii, nicht aber A. comandoni, A. culbertsoni oder Vermamoeba vermiformis .[57]
- Spezies Megavirus baoshanense mit
- Gattung Megavirus (Mimiviridae: Gruppe I Linie C, MC,[21] Courdo11-Linie oder Megavirus-Linie)
T Bei Namensgleichheit mit der Spezies deutet ein hochgestelltes ‚T‘ einen Typus- oder Referenzstamm an.
Die Phylogenie der Mimiviridae-Gruppe I, d. h. der Unterfamilie Megamimivirinae inkl. der Gattung Mimivirus (Mimiviren s. l.) ist nach Aylward et al. (2021) vereinfacht wie folgt:[5]
| Mimiviridae-Gruppe I |
| ||||||||||||||||||||||||||||||||||||||||||
Frühere Analysen wie die von Abrahão et al. (2018) hatten für die innere Phylogenie der Gattung Megavirus noch ein etwas anderes Bild abgegeben als es der aktuellen Aufteilung der Stämme auf die drei offiziellen Megavirus-Spezies entspricht.[42]
Literatur
- Adam Monier, Jean-Michel Claverie, Hiroyuki Ogata: Taxonomic distribution of large DNA viruses in the sea. In: Genome Biology. Band 9, Nr. 7, 2008, S. R106, doi:10.1186/gb-2008-9-7-r106, PMID 18598358, PMC 2530865 (freier Volltext).
- Elodie Ghedin, Jean-Michel Claverie: Mimivirus relatives in the Sargasso sea. In: Virology Journal. Band 2, 2005, S. 62, doi:10.1186/1743-422X-2-62, PMID 16105173, PMC 1215527 (freier Volltext).
- James L. Van Etten: Giant Viruses. In: American Scientist. Band 99, Nr. 4, 2011, S. 304, doi:10.1511/2011.91.304.
Weblinks
- Stuart Siddell: Why virus taxonomy is important, Microbiology Society, 13. Februar 2018
- Welcome to the Giant Virus site, home of mimivirus and other large DNA viruses. GiantVirus.org – eine Informationsquelle über das Genom von Riesenviren.
- Jean-Luc Goudet: Nouméavirus, un étonnant virus géant qui agit à distance. Futura Santé, 1. Mai 2017 (mit bildlichen Darstellungen von Megavirus, französisch)
Anmerkungen
- ↑ Die zytoplasmatischen Seeds sind daher Vorstufen der Virus-Fabriken und enthalten nicht viel mehr als das Virus-Genom.
Einzelnachweise
- ↑ a b c d e f g h ICTV: Master Species Lists § ICTV Master Species List 2022 MSL38 v1 (xlsx), 8. April 2023.
- ↑ a b c Frederik Schulz, Lauren Alteio, Danielle Goudeau, Elizabeth M. Ryan, Feiqiao B. Yu, Rex R. Malmstrom, Jeffrey Blanchard, Tanja Woyke: Hidden diversity of soil giant viruses. In: Nature Communications, Band 9, Nr. 4881, 19. November 2018, doi:10.1038/s41467-018-07335-2.
- ↑ a b c d e f ICTV: ICTV Taxonomy history: Acanthamoeba polyphaga mimivirus, EC 51, Berlin, Germany, July 2019; Email ratification March 2020 (MSL #35)
- ↑ a b c Defne Arslan, Matthieu Legendre, Virginie Seltzer, Chantal Abergel, Jean-Michel Claverie: Distant Mimivirus relative with a larger genome highlights the fundamental features of Megaviridae. In: PNAS. 10. Oktober 2011, doi:10.1073/pnas.1110889108.
- ↑ a b c d e f g Frank O. Aylward, Jônatas S. Abrahão, Corina P. D. Brussaard C, Matthias G. Fischer, Mohammad Moniruzzaman, Hiroyuki Ogata, Curtis A. Suttle: Create 3 new families, 3 subfamilies, 13 genera, and 20 new species within the order Imitervirales (phylum Nucleocytoviricota) and rename two existing species (zip:docx). Vorschlag 2022.004F an das ICTV vom Oktober 2021.
Anm.: Entgegen Vorschlag (Tbl. 1) wurde die Gattung Mimivirus nicht in die Unterfamilie Megamimivirinae aufgenommen. Phylogenetische Analysen zeigten zu deren Mitgliedern einen größeren Abstand als diese untereinander. - ↑ Didier Raoult, Stéphane Audic, Catherine Robert, Chantal Abergel, Patricia Renesto, Hiroyuki Ogata, Bernard La Scola, Marie Suzan, Jean-Michel Claverie: The 1.2-Megabase Genome Sequence of Mimivirus. In: Science. Band 306, Nr. 5700, 19. November 2004, S. 1344–1350, doi:10.1126/science.1101485, PMID 15486256.
- ↑ a b Christoph M. Deeg, Cheryl-Emiliane T. Chow, Curtis A. Suttle: The kinetoplastid-infecting Bodo saltans virus (BsV), a window into the most abundant giant viruses in the sea… In: eLife Sciences, 7, März 2018, doi:10.7554/eLife.33014.
- ↑ List of the main “giant” viruses known as of today (March 2019) (PDF) Centre national de la recherche scientifique, Université Aix Marseille, März 2019.
- ↑ a b Morgan Gaia, Isabelle Pagnier, Angélique Campocasso, Ghislain Fournous, Didier Raoult, Bernard La Scola: Broad spectrum of mimiviridae virophage allows its isolation using a mimivirus reporter. PLoS One, 2013, 8, S. e61912, PMC 3626643 (freier Volltext), doi:10.1371/journal.pone.0061912.
- ↑ a b Anthony Levasseur, Meriem Bekliz, Eric Chabrière, Pierre Pontarotti, Bernard La Scola, Didier Raoult: MIMIVIRE is a defence system in mimivirus that confers resistance to virophage. In: Nature, 2016, doi:10.1038/nature17146, PMID 26934229, ResearchGate. Siehe insbes.:
- Extended Data Extended Data Fig. 1: Histogram depicting the replication of Zamilon and Sputnik 3 DNA in Mimiviridae after its phylogenetic classification into lineages A, B and C.
- Supplementary Information. (Memento des vom 3. August 2019 im Internet Archive) Info: Der Archivlink wurde automatisch eingesetzt und noch nicht geprüft. Bitte prüfe Original- und Archivlink gemäß Anleitung und entferne dann diesen Hinweis. (PDF)
- ↑ Ewen Callaway: CRISPR-like 'immune' system discovered in giant virus. In: Nature: News, 29. Februar 2016; doi:10.1038/nature.2016.19462.
- ↑ T. J. Rowbotham: Isolation of Legionella pneumophila from clinical specimens via amoebae, and the interaction of those and other isolates with amoebae. In: Journal of Clinical Pathology. Band 36, Nr. 9, 1983, S. 978–986, doi:10.1136/jcp.36.9.978, PMID 6350372, PMC 498455 (freier Volltext).
- ↑ Autor=Nathan Zauberman, Y. Mutsafi, D. B. Halevy, E. Shimoni, E. Klein, C. Xiao, S. Sun, A. Minsky: Distinct DNA exit and packaging portals in the virus Acanthamoeba polyphaga mimivirus. In: PLoS Biol. Band 6, Nr. 5, 2008, S. e114, doi:10.1371/journal.pbio.0060114, PMID 18479185, PMC 2430901 (freier Volltext).
- ↑ David M. Needham, Susumu Yoshizawa, Toshiaki Hosaka, Camille Poirier, Chang Jae Choi, Elisabeth Hehenberger, Nicholas A. T. Irwin, Susanne Wilken, Cheuk-Man Yung, Charles Bachy, Rika Kurihara, Yu Nakajima, Keiichi Kojima, Tomomi Kimura-Someya, Guy Leonard, Rex R. Malmstrom, Daniel R. Mende, Daniel K. Olson, Yuki Sudo, Sebastian Sudek, Thomas A. Richards, Edward F. DeLong, Patrick J. Keeling, Alyson E. Santoro, Mikako Shirouzu, Wataru Iwasaki, Alexandra Z. Worden: A distinct lineage of giant viruses brings a rhodopsin photosystem to unicellular marine predators. In: PNAS, 23. September 2019, ISSN 0027-8424; doi:10.1073/pnas.1907517116, inklusive Supplement 1 (xlsx).
- ↑ Hiroyuki Ogata, J. Ray, K. Toyoda, R. A. Sandaa, K. Nagasaki, G. Bratbak, Jean-Michel Claverie: Two new subfamilies of DNA mismatch repair proteins (MutS) specifically abundant in the marine environment. In: The ISME Journal. Band 5, Nr. 7, 2011, S. 1143–1151, doi:10.1038/ismej.2010.210, PMID 21248859, PMC 3146287 (freier Volltext).
- ↑ Jean-Michel Claverie, Chantal Abergel: Mimivirus: The emerging paradox of quasi-autonomous viruses. In: Trends in Genetics. Band 26, Nr. 10, 2010, S. 431–437, doi:10.1016/j.tig.2010.07.003, PMID 20696492.
- ↑ Matthieu Legendre, Defne Arslan, Chantal Abergel, Jean-Michel Claverie: Genomics of Megavirus and the elusive fourth domain of Life. In: Commun Integr Biol. Band 5, Nr. 1, 1. Januar 2012, S. 102–106; doi:10.4161/cib.18624, PMID 22482024, PMC 3291303 (freier Volltext).
- ↑ Gwyneth Dickey Zakaib: The challenge of microbial diversity: Out on a limb. In: Nature. Band 476, Nr. 7358, 2011, S. 20–21, doi:10.1038/476020a, PMID 21814255.
- ↑ Natalya Yutin, Eugene V. Koonin: Hidden evolutionary complexity of Nucleo-Cytoplasmic Large DNA viruses of eukaryotes. In: Virology Journal, Band 9, Nr. 161, 14. August 2012; doi:10.1186/1743-422X-9-161.
- ↑ NCBI Taxonomy Browser: Search for …, Nucleotide: Search.
- ↑ a b c Sandra Jeudy, Lionel Bertaux, Jean-Marie Alempic, Audrey Lartigue, Matthieu Legendre, Lucid Belmudes, Sébastien Santini, Nadège Philippe, Laure Beucher, Emanuele G. Biondi, Sissel Juul, Daniel J. Turner, Yohann Couté, Jean-Michel Claverie, Chantal Abergel: Exploration of the propagation of transpovirons within Mimiviridae reveals a unique example of commensalism in the viral world. In: The ISME Journal, Band 14, S. 727–739, 10. Dezember 2019; doi:10.1038/s41396-019-0565-y, PMID 31822788, PMC 7031253 (freier Volltext).
- ↑ a b c Sailen Barik: A Family of Novel Cyclophilins, Conserved in the Mimivirus Genus of the Giant DNA Viruses. In: Computational and Structural Biotechnology Journal, Band 16, Juli 2018, S. 231–236, doi:10.1016/j.csbj.2018.07.001
- ↑ a b Julien Guglielmini, Anthony C. Woo, Mart Krupovic, Patrick Forterre, Morgan Gaia: pnas.org In: PNAS, Band 116, Nr. 39, 10./24. September 2019, S. 19585–19592, doi:10.1073/pnas.1912006116, PMID 31506349, Fig. 2.
- ↑ NCBI Taxonomy Browser: Megavirus chiliensis, Details: Megavirus chiliensis (species).
- ↑ NCBI Taxonomy Browser: Megavirus lba (no rank), Nucleotide: Megavirus lba isolate LBA111, …
- ↑ a b Hanene Saadi, Dorine-Gaelle Ikanga Reteno, Philippe Colson, Sarah Aherfi, Philippe Minodier, Isabelle Pagnier, Didier Raoult, Bernard La Scola: Shan Virus: A New Mimivirus Isolated from the Stool of a Tunisian Patient with Pneumonia. In: Intervirology, 2013, Nr. 56, S. 424–429, doi:10.1159/000354564.
- ↑ a b c d e f g h i j k l m n o p q Didier Raoult, Anthony Levasseur, Bernard La Scola: PCR Detection of Mimivirus. In: Emerging Infectious Diseases, Juni 2017, Band 23, Nr. 6, S. 1044–1045; doi:10.3201/eid2306.161896, PDF.
- ↑ a b c Morgan Gaia, Samia Benamar, Mondher Boughalmi, Isabelle Pagnier, Olivier Croce, Philippe Colson, Didier Raoult, Bernard La Scola: Zamilon, a Novel Virophage with Mimiviridae Host Specificity. In: PLoS One. 9. Jahrgang, Nr. 4, 2014, S. e94923, doi:10.1371/journal.pone.0094923, PMID 24747414, PMC 3991649 (freier Volltext) – (englisch). Online veröffentlicht am 18. April 2014.
- ↑ NCBI Taxonomy Browser: Megavirus vitis (species).
- ↑ a b c d Julien Andreani, Jacques Y. B. Khalil, Emeline Baptiste, Issam Hasni, Caroline Michelle, Didier Raoult, Anthony Levasseur, Bernard La Scola: Orpheovirus IHUMI-LCC2: A New Virus among the Giant Viruses. In: Front. Microbiol., 22. Januar 2018, doi:10.3389/fmicb.2017.02643.
- ↑ NCBI Taxonomy Browser: Courdo virus CE11 (species), Nucleotide: Courdo virus CE11 …
- ↑ Clara Rolland, Julien Andreani, Amina Cherif Louazani, Sarah Aherfi, Rania Francis, Rodrigo Rodrigues, Ludmila Santos Silva, Dehia Sahmi, Said Mougari, Nisrine Chelkha, Meriem Bekliz, Lorena Silva, Felipe Assis, Fábio Dornas, Jacques Yaacoub Bou Khalil, Isabelle Pagnier, Christelle Desnues, Anthony Levasseur, Philippe Colson, Jônatas Abrahão, Bernard La Scola: Discovery and Further Studies on Giant Viruses at the IHU Mediterranee Infection That Modified the Perception of the Virosphere. In: Viruses, Band 11, Nr. 4, März/April 2019, pii: E312; doi:10.3390/v11040312, PMID 30935049, PMC 6520786 (freier Volltext). Siehe insbes. Tab. 1.
- ↑ NCBI Taxonomy Browser: Megavirus courdo5 (species).
- ↑ NCBI Taxonomy Browser: Megavirus courdo7 (species).
- ↑ a b Hansika Chhabra: Giant viruses found in water samples from Mumbai. Hindu BusinessLine: Science, Bangalore, 9. Mai 2019.
- ↑ Anirvan Chatterjee, Farhan Ali, Disha Bange, Kiran Kondabagil: Complete Genome Sequence of a New Megavirus Family Member Isolated from an Inland Water Lake for the First Time in India. In: ASM Journals: Microbiology Resource Announcements, Band 4, Nr. 3, 16. Juni 2016, doi:10.1128/genomeA.00402-16.
- ↑ Anirvan Chatterjee, Thomas Sicheritz-Pontén, Rajesh Yadav, Kiran Kondabagil: Genomic and metagenomic signatures of giant viruses are ubiquitous in water samples from sewage, inland lake, waste water treatment plant, and municipal water supply in Mumbai, India. In: Scientific Reports, Band 9, Nr. 3690, 6. März 2019; doi:10.1038/s41598-019-40171-y, PMID 30842490, PMC 6403294 (freier Volltext).
- ↑ NCBI Taxonomy Browser: Megavirus avenue9 (species).
- ↑ NCBI Taxonomy Browser: Megavirus balcon (species).
- ↑ NCBI Taxonomy Browser: Megavirus battle43 (species).
- ↑ NCBI Taxonomy Browser: Megavirus bus (species).
- ↑ a b Jônatas Abrahão, Lorena Silva, Ludmila Santos Silva, Jacques Yaacoub Bou Khalil, Rodrigo Rodrigues, Thalita Arantes, Felipe Assis, Paulo Boratto, Miguel Andrade, Erna Geessien Kroon, Bergmann Ribeiro, Ivan Bergier, Herve Seligmann, Eric Ghigo, Philippe Colson, Anthony Levasseur, Guido Kroemer, Didier Raoult, Bernard La Scola: Tailed giant Tupanvirus possesses the most complete translational apparatus of the known virosphere. In: Nature Communications. 9. Jahrgang, Nr. 1, 27. Februar 2018, doi:10.1038/s41467-018-03168-1 (englisch).
- ↑ Anirvan Chatterjee, Thomas Sicheritz-Pontén, Rajesh Yadav, Kiran Kondabagil: Isolation and complete genome sequencing of Mimivirus bombay, a Giant Virus in sewage of Mumbai, India. In: Genomics Data, Band 9, Nr. C, Mai 2016; doi:10.1016/j.gdata.2016.05.013, ResearchGate. Siehe insbes. Fig. 2.
- ↑ NCBI Taxonomy Browser: Megavirus feuillage (species).
- ↑ NCBI Taxonomy Browser: Megavirus J3 (species).
- ↑ Jean-Marie Alempic, Audrey Lartigue, Artemiy E. Goncharov, Guido Grosse, Jens Strauss, Alexey N. Tikhonov, Alexander N. Fedorov, Olivier Poirot, Matthieu Legendre, Sébastien Santini, Chantal Abergel, Jean-Michel Claverie: An Update on Eukaryotic Viruses Revived from Ancient Permafrost . In: MDPI: Viruses, Band 15, Nr. 2, S. 564, Special Issue Research Progresses of Giant Viruses; doi:10.3390/v15020564. Dazu:
- Chris Panella, Morgan McFall-Johnsen (Business Insider): Ancient 'Zombie' Virus Revived Almost 50,000 Years Later Is Still Infectious. Auf: sciencealert vom 10. März 2023.
- ↑ NCBI Taxonomy Browser: Megavirus mont1 (species).
- ↑ NCBI Taxonomy Browser: Megavirus montpellier (species).
- ↑ NCBI Taxonomy Browser: Megavirus potager (species).
- ↑ NCBI Taxonomy Browser: Megavirus ursino (species).
- ↑ NCBI Taxonomy Browser: Megavirus shan (species).
- ↑ NCBI Taxonomy Browser: Megavirus T1 (species).
- ↑ NCBI Taxonomy Browser: Megavirus T4 (species).
- ↑ NCBI Taxonomy Browser: Megavirus T6 (species).
- ↑ NCBI Taxonomy Browser: Bandra megavirus (species).
- ↑ NCBI Taxonomy Browser: Afrovirus urmite69 (species).
- ↑ Haruna Takahashi, Sho Fukaya, Chihong Song, Kazuyoshi Murata, Masaharu Takemura: Morphological and Taxonomic Properties of the Newly Isolated Cotonvirus japonicus, a New Lineage of the Subfamily Megavirinae. In: ASM Journals: Journal of Virology, Band 95, Nr. 18, 25. August 2021; doi:10.1128/JVI.00919, ResearchGate.
Auf dieser Seite verwendete Medien
Autor/Urheber: Morgan Gaia, Samia Benamar, Mondher Boughalmi, Isabelle Pagnier, Olivier Croce, Philippe Colson, Didier Raoult and Bernard La Scola, Lizenz: CC BY 4.0
Virus factory in amoeba co-infected with Zamilon and Mont1, showing abnormal Mont1 particles (arrows) (scale bar 0.1 µm)
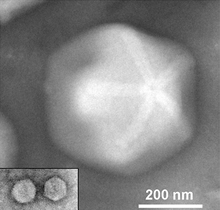
Autor/Urheber: Sarah Duponchel and Matthias G. Fischer, Lizenz: CC BY-SA 4.0
Electron microscope images depicting capsids shape of the giant virus Megavirus vitis (with a visible stargate structure) and its associated virophage Zamilon vitis (inset).
Negative stain EM courtesy of C. Abergel, Aix-Marseille Université, France.
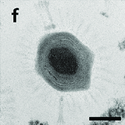
Autor/Urheber: Chantal Abergel, Lizenz: CC BY-SA 3.0
Megavirus particle. Thin section, electron microscopy
Autor/Urheber: Haruna Takahashi, Sho Fukaya,Chihong Song, Kazuyoshi Murata, Masaharu Takemura, Lizenz: CC BY 4.0
Electron micrographs of cotonvirus and related particles. TEM image of Megavirus musashi (lineage C) particles. Scale bars: 200 nm.
Autor/Urheber: Morgan Gaia, Isabelle Pagnier, Angélique Campocasso, Ghislain Fournous, Didier Raoult, Bernard La Scola, Lizenz: CC BY-SA 4.0
Aspect of Sputnik 3 virophage produced in virus factory of Courdo11 (Mimivirus group C – scale bar 2 µm)