Lysin
| Strukturformel | |||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
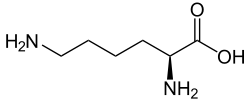 | |||||||||||||||||||
| Strukturformel des natürlich vorkommenden L-Lysins | |||||||||||||||||||
| Allgemeines | |||||||||||||||||||
| Name | Lysin | ||||||||||||||||||
| Andere Namen |
| ||||||||||||||||||
| Summenformel | C6H14N2O2 | ||||||||||||||||||
| Kurzbeschreibung | farblose Nadeln oder hexagonale Plättchen[1] | ||||||||||||||||||
| Externe Identifikatoren/Datenbanken | |||||||||||||||||||
| |||||||||||||||||||
| Arzneistoffangaben | |||||||||||||||||||
| ATC-Code | |||||||||||||||||||
| Eigenschaften | |||||||||||||||||||
| Molare Masse | 146,19 g·mol−1 | ||||||||||||||||||
| Aggregatzustand | fest | ||||||||||||||||||
| Schmelzpunkt |
| ||||||||||||||||||
| pKS-Wert | |||||||||||||||||||
| Sicherheitshinweise | |||||||||||||||||||
| |||||||||||||||||||
| Wenn nicht anders vermerkt, gelten die angegebenen Daten bei Standardbedingungen (0 °C, 1000 hPa). | |||||||||||||||||||
Lysin, abgekürzt Lys oder K, ist in seiner natürlichen L-Form eine essentielle proteinogene α-Aminosäure. Es ist benannt nach der Isolierung aus aufgelöstem Casein, von λύσις lysis „Auflösung“.[5]
Der Einbuchstabencode K für Lysin wurde aufgrund der alphabetischen Nähe zugeordnet, nachdem L bereits dem strukturell einfacheren Leucin und M dem Methionin zugeordnet war.[6][7]
Stereoisomerie
In den Proteinen kommt, neben anderen Aminosäuren, ausschließlich L-Lysin [Synonym: (S)-Lysin] peptidisch gebunden vor. Enantiomer dazu ist das spiegelbildliche D-Lysin [Synonym: (R)-Lysin], das in Proteinen nicht vorkommt. Racemisches DL-Lysin [Synonyme: (RS)-Lysin und (±)-Lysin] hat eine geringere Bedeutung als L-Lysin, besitzt jedoch kommerzielle Bedeutung als basische Komponente in Arzneistoff-Salzen, z. B. mit Acetylsalicylsäure.
Wenn in diesem Text oder in der wissenschaftlichen Literatur „Lysin“ ohne weiteren Namenszusatz erwähnt wird, ist L-Lysin gemeint.
| Isomere von Lysin | ||
| Name | L-Lysin | D-Lysin |
| Andere Namen | (S)-Lysin | (R)-Lysin |
| Strukturformel |  |  |
| CAS-Nummer | 56-87-1 | 923-27-3 |
| 70-54-2 (DL) | ||
| EG-Nummer | 200-294-2 | 213-091-9 |
| 200-740-6 (DL) | ||
| ECHA-Infocard | 100.000.268 | 100.011.902 |
| 100.000.673 (DL) | ||
| PubChem | 5962 | 57449 |
| 866 (DL) | ||
| DrugBank | DB00123 | - |
| - (DL) | ||
| FL-Nummer | 17.026 | - |
| 17.013 (DL) | ||
| Wikidata | Q20816880 | Q27077084 |
| Q178430 (DL) | ||
Geschichte
Nach der Entdeckung des Phenylalanins äußerte der deutsche Chemiker Ernst Schulze die Vermutung, dass Proteine noch aus weiteren Aminosäuren als den bis dahin bekannten aufgebaut sind. Unter anderem dies veranlasste Edmund Drechsel, erneut die Bestandteile der Salzsäurespaltung von Casein zu untersuchen.[8] Nach der Behandlung mit Phosphorwolframsäure gelang es Drechsel 1889 die Platinsalze von Lysin zu isolieren.[9] Sein Schüler Max Siegfried veröffentlichte 1891 die richtige Zusammensetzung dieser Aminosäure.[10] 1902 gelang dem Nobelpreisträger Emil Fischer und seinen Assistenten Fritz Weigert die finale Aufklärung der Struktur mit der Synthese von Lysin.[11]
Eigenschaften

Gemeinsam mit L-Arginin und L-Histidin gehört L-Lysin in die Gruppe der basischen und zugleich proteinogenen α-Aminosäuren oder Hexonbasen. Lysin besitzt zwei basische primäre Aminogruppen, eine in α-Position zur Carboxygruppe und eine in der ε-Position der Seitenkette. Die Ladung des Lysins ist – wie bei allen Aminosäuren – vom pH-Wert abhängig. Lysin liegt überwiegend als „inneres Salz“ bzw. Zwitterion vor, dessen Bildung dadurch zu erklären ist, dass das Proton der Carboxygruppe zum freien Elektronenpaar des Stickstoffatoms der ε-Aminogruppe wandert, die stärker basisch ist als die α-Aminogruppe:[12]
Im elektrischen Feld wandert das Zwitterion nicht, da es als Ganzes ungeladen ist. Genau genommen ist dies am isoelektrischen Punkt (bei einem bestimmten pH-Wert, hier 9,82[13]) der Fall, bei dem das Lysin auch seine geringste Löslichkeit in Wasser besitzt.
Weitere physikochemische Daten für Lysin sind:[14]
- isoelektrischer Punkt: 9,74
- Van-der-Waals-Volumen: 135 Å3
- Hydrophobizitätsgrad: −3,9
Industrielle Herstellung
Industriell werden mehrere 100.000 Tonnen L-Lysin pro Jahr hergestellt. L-Lysin wird heute ausschließlich nach der Fermentationsmethode hergestellt, obgleich organisch-chemische Syntheserouten entwickelt wurden.[15]
Vorkommen
Lysin ist eine für den Menschen und andere Säugetiere, wie beispielsweise Schweine, essentielle Aminosäure und muss mit der Nahrung zugeführt werden. Die folgenden Beispiele für den Gehalt an Lysin beziehen sich jeweils auf 100 g des Lebensmittels, zusätzlich ist der prozentuale Anteil am Gesamtprotein angegeben:[16][17]

| Lebensmittel | Gesamtprotein | Lysin | Anteil |
|---|---|---|---|
| Rindfleisch, roh | 21,26 g | 1,797 g | 8,5 % |
| Hähnchenbrustfilet, roh | 23,09 g | 1,962 g | 8,5 % |
| Kürbiskern | 35,49 g | 2,283 g | 6,4 % |
| Lachs, roh | 20,42 g | 1,870 g | 9,2 % |
| Erbsen, getrocknet | 24,55 g | 1,772 g | 7,2 % |
| Tofu, fest | 15,51 g | 1,000 g | 6,4 % |
| Hühnerei | 12,58 g | 0,914 g | 7,3 % |
| Kuhmilch, 3,7 % Fett | 3,28 g | 0,260 g | 7,9 % |
| Walnüsse | 15,23 g | 0,424 g | 2,8 % |
| Weizen-Vollkornmehl | 13,70 g | 0,378 g | 2,8 % |
| Mais-Vollkornmehl | 6,93 g | 0,195 g | 2,8 % |
| Gerste | 8–12 g | 0,446 g | 4–6 % |
| Reis, ungeschält | 7,94 g | 0,303 g | 3,8 % |
| Buchweizen-Mehl | 11,73 g | 0,595 g | 5,1 % |
| Quinoa | 13 g | 0,860 g | 6,6 % |
Alle diese Nahrungsmittel enthalten praktisch ausschließlich chemisch gebundenes L-Lysin als Proteinbestandteil, jedoch kein freies L-Lysin. Getreide enthalten meist geringere L-Lysin-Anteile unter den Aminosäuren des Proteinbestandteils, als es für die menschliche Ernährung optimal ist.[19]
Die Einschätzungen des Tagesbedarfs für gesunde Erwachsene reichen, je nach verwendeter Methode, von 8 bis 45 mg Lysin pro Kilogramm Körpergewicht. Eine Expertenkommission der FAO/WHO/UNU ging im Jahr 2002 von einem täglichen Bedarf zwischen 30 mg und 64 mg pro Kilogramm Körpergewicht für Säuglinge und Erwachsene aus.[20]
Funktionen

Lysin ist eine der Aminosäuren, die bevorzugt posttranslational modifiziert werden. Dabei kann die positive Ladung erhalten bleiben (mono- und di-Methylierung), verschwinden (Acetylierung) oder negativ werden (Malonylierung, Succinylierung, Glutarylierung). Im Kollagen wurde ein modifiziertes Lysin gefunden, das Hydroxylysin mit einer OH-Gruppe in der Seitenkette, katalysiert unter Mitwirkung des Enzyms Lysylhydroxylase und des Cofaktors Ascorbinsäure (Vitamin C). Hydroxylysin erlaubt die nachfolgende O-Glykosylierung des Kollagenmoleküls im Endoplasmatischen Retikulum und Golgi-Apparat. Die Glykosylierung bestimmt die Packungsdichte dieses wichtigen Bindegewebeproteins und wird auch mit der Steuerung der Kollagenabgabe aus der Zelle (Exozytose) in Verbindung gebracht.
Eine herausragende Bedeutung haben die Aminosäure Lysin und ihre methylierten Formen bei der Histonmodifikation. Speziell die Lysin-Reste am Histon 3 und 4 sind die bevorzugten Substrate von Histon-modifizierenden Enzymen (Histon-Acetyltransferasen, Histon-Deacetylasen, Histon-Methyltransferasen, Histon-Demethylasen). Durch die dadurch erfolgte Lysin-Acetylierung oder Lysin-Methylierung (oft an mehreren Stellen im Molekül) ändert sich die elektrostatische Wechselwirkung zwischen den Histonen und der genomischen DNA, wodurch bei letzterer der Zugang von Transkriptionsfaktoren und DNA-modifizierenden Enzymen reguliert wird. Diese Histon-Lysin-Modifikationen sind damit ein wesentlicher, molekularer Mechanismus der epigenetischen Gen-Regulation.
Eine weitere Modifikation ist die Ubiquitinierung in Proteinen, die damit für den Abbau durch das Proteasom markiert werden.
Beim Abbau des Lysins (Eiweißfäule) entsteht durch Decarboxylierung das Leichengift Cadaverin.
Biosynthese

In der Natur wurden zwei Wege für die Synthese von Lysin identifiziert. Der Diaminopimelat (DAP)-Weg gehört zur Familie der von Aspartat abgeleiteten Biosynthese, die auch an der Synthese von Threonin, Methionin und Isoleucin beteiligt ist.[21][22] Der α-Aminoadipat (AAA)-Weg hingegen ist Teil der Glutamat-Biosynthese-Familie.[23][24]
Der DAP-Weg findet sich sowohl in Prokaryoten als auch in Pflanzen und beginnt mit der durch Dihydrodipicolinat-Synthase (DHDPS) (E.C 4.3.3.7) katalysierten Kondensationsreaktion zwischen dem von Aspartat abgeleiteten L-Aspartat-Semialdehyd und Pyruvat zur Bildung von (4S)-4-Hydroxy- 2,3,4,5-Tetrahydro-(2S)-dipicolinsäure (HTPA).[25][26][27][28][29] Das Produkt wird dann durch Dihydrodipicolinat-Reduktase (DHDPR) (E.C 1.3.1.26) mit NAD(P)H als Protonendonor reduziert, um 2,3,4,5-Tetrahydrodipicolinat (THDP) zu erhalten.[30]
Verwendung
Die Hauptmengen des industriell erzeugten L-Lysins werden in der Futtermittelsupplementierung eingesetzt, um den Nährwert natürlicher Futtermittel (Getreide) mit einem geringen Gehalt an L-Lysin deutlich zu steigern.[15][31]
Racemisches DL-Lysin besitzt kommerzielle Bedeutung als basische Komponente in Arzneistoff-Salzen, z. B. mit Acetylsalicylsäure (ASS).
L-Lysin ist Bestandteil von Infusionslösungen zur parenteralen Ernährung und zur Behandlung hypochlorämischer Alkalosen.[32]
Lysin wird auch zur Wirkbeschleunigung bei schmerzhemmenden Mitteln verwendet, insbesondere in Verbindung mit Ibuprofen.
Biochemie
Für detaillierte Strukturformeln siehe auch Abschnitt Weblinks
L-Lysin kann in zwei Moleküle Acetyl-CoA abgebaut werden.
Weiterführende Literatur
- D. Datta, A. Bhinge, V. Chandran: Lysine: Is it worth more? In: Cytotechnology. Band 36, Nummer 1–3, Juli 2001, S. 3–32, doi:10.1023/A:1014097121364. PMID 19003311. PMC 3449675 (freier Volltext).
Weblinks
Einzelnachweise
- ↑ a b c d e f Eintrag zu L-Lysin. In: Römpp Online. Georg Thieme Verlag, abgerufen am 29. Mai 2014.
- ↑ The Merck Index. An Encyclopaedia of Chemicals, Drugs and Biologicals. 14. Auflage. 2006, ISBN 0-911910-00-X, S. 698.
- ↑ a b J. C. Eck, C. S. Marvel: dl-Lysine Hydrochlorides In: Organic Syntheses. 19, 1939, S. 61, doi:10.15227/orgsyn.019.0061; Coll. Vol. 2, 1943, S. 374 (PDF).
- ↑ a b Datenblatt Lysin bei Sigma-Aldrich, abgerufen am 12. Juni 2011 (PDF).
- ↑ Hubert Bradford. Vickery, Carl L. A. Schmidt: The History of the Discovery of the Amino Acids. In: Chemical Reviews. Band 9, Nr. 2, 1. Oktober 1931, S. 169–318, doi:10.1021/cr60033a001.
- ↑ IUPAC-IUB Commission on Biochemical Nomenclature A One-Letter Notation for Amino Acid Sequences. In: Journal of Biological Chemistry. Band 243, Nr. 13, 10. Juli 1968, S. 3557–3559, doi:10.1016/S0021-9258(19)34176-6 (englisch, jbc.org [PDF]).
- ↑ M. Saffran: Amino acid names and parlor games: from trivial names to a one-letter code, amino acid names have strained students' memories. Is a more rational nomenclature possible? In: Biochemical Education. Band 26, Nr. 2, April 1998, S. 116–118, doi:10.1016/S0307-4412(97)00167-2 (englisch, elsevier.com).
- ↑ Sabine Hansen:Die Entdeckung der proteinogenen Aminosäuren von 1805 in Paris bis 1935 in Illinois. ( vom 15. Juni 2016 im Internet Archive) Berlin 2015.
- ↑ E. Drechsel: Zur Kenntniss der Spaltungsprodukte des Caseins. In: Journal für Praktische Chemie. Band 39, 1889, S. 425ff, doi:10.1002/prac.18890390135.
- ↑ M. Siegfried: Zur Kenntnis der Spaltungsprodukte der Eiweisskörper. In: Ber Deutschen Chem Ges. Band 24, 1891, S. 418ff.
- ↑ E. Fischer, F. Weigert: Synthese der α, ϵ-Diaminocapronsäure (Inactives Lysin). In: Berichte der Deutschen Chemischen Gesellschaft. Band 35, Nr. 3, 1902, S. 3772ff.
- ↑ Hans-Dieter Jakubke, Hans Jeschkeit: Aminosäuren, Peptide, Proteine. Verlag Chemie, 1982, ISBN 3-527-25892-2, S. 41.
- ↑ P. M. Hardy: The Protein Amino Acids. In: G. C. Barrett (Hrsg.): Chemistry and Biochemistry of the Amino Acids. Chapman and Hall, 1985, ISBN 0-412-23410-6, S. 9.
- ↑ Paul G. Higgs, Teresa K. Attwood: Bioinformatics and Molecular Evolution. John Wiley & Sons, 2009, ISBN 978-1-4443-1118-1, S. 24 (eingeschränkte Vorschau in der Google-Buchsuche).
- ↑ a b Y. Izumi u. a.: Herstellung und Verwendung von Aminosäuren. In: Angewandte Chemie. Band 90, 1978, S. 187–194. doi:10.1002/ange.19780900307
- ↑ Nährstoffdatenbank des US-Landwirtschaftsministeriums, 21. Auflage.
- ↑ Tofu fest
- ↑ Jesse P. Greenstein, Milton Winitz: Chemistry of the Amino Acids. Robert E. Krieger Publishing, Malabar (Florida) 1961, ISBN 0-89874-484-9, S. 4.
- ↑ G. C. Barrett: Chemistry and Biochemistry of the Amino Acids. Chapman and Hall, London / New York 1985, ISBN 0-412-23410-6, S. 12.
- ↑ D. Tomé, C. Bos: Lysine requirement through the human life cycle. Band 137, 2007, S. 1642S–1645S. PMID 17513440.
- ↑ A. O. Hudson, C. Bless, P. Macedo, S. P. Chatterjee, B. K. Singh, C. Gilvarg, T. Leustek: Biosynthesis of lysine in plants: evidence for a variant of the known bacterial pathways. In: Biochimica et Biophysica Acta (BBA) - General Subjects. Band 1721, Nr. 1–3, Januar 2005, S. 27–36, doi:10.1016/j.bbagen.2004.09.008, PMID 15652176 (englisch).
- ↑ A. M. Velasco, J. I. Leguina, A. Lazcano: Molecular evolution of the lysine biosynthetic pathways. In: Journal of Molecular Evolution. Band 55, Nr. 4, Oktober 2002, S. 445–459, doi:10.1007/s00239-002-2340-2, PMID 12355264, bibcode:2002JMolE..55..445V (englisch).
- ↑ T. Miyazaki, J. Miyazaki, H. Yamane, M. Nishiyama: alpha-Aminoadipate aminotransferase from an extremely thermophilic bacterium, Thermus thermophilus. In: Microbiology. Band 150, Pt 7, Juli 2004, S. 2327–2334, doi:10.1099/mic.0.27037-0, PMID 15256574 (englisch).
- ↑ H. Xu, B. Andi, J. Qian, A. H. West, P. F. Cook: The alpha-aminoadipate pathway for lysine biosynthesis in fungi. In: Cell Biochemistry and Biophysics. Band 46, Nr. 1, 2006, S. 43–64, doi:10.1385/CBB:46:1:43, PMID 16943623 (englisch).
- ↑ S. C. Atkinson, C. Dogovski, M. T. Downton, P. E. Czabotar, R. C. Dobson, J. A. Gerrard, J. Wagner, M. A. Perugini: Structural, kinetic and computational investigation of Vitis vinifera DHDPS reveals new insight into the mechanism of lysine-mediated allosteric inhibition. In: Plant Molecular Biology. Band 81, Nr. 4–5, März 2013, S. 431–446, doi:10.1007/s11103-013-0014-7, PMID 23354837 (englisch).
- ↑ M. D. Griffin, J. M. Billakanti, A. Wason, S. Keller, H. D. Mertens, S. C. Atkinson, R. C. Dobson, M. A. Perugini, J. A. Gerrard, F. G. Pearce: Characterisation of the first enzymes committed to lysine biosynthesis in Arabidopsis thaliana. In: PLOS ONE. Band 7, Nr. 7, 2012, S. e40318, doi:10.1371/journal.pone.0040318, PMID 22792278, PMC 3390394 (freier Volltext), bibcode:2012PLoSO...740318G (englisch).
- ↑ T. P. Soares da Costa, A. C. Muscroft-Taylor, R. C. Dobson, S. R. Devenish, G. B. Jameson, J. A. Gerrard: How essential is the 'essential' active-site lysine in dihydrodipicolinate synthase? In: Biochimie. Band 92, Nr. 7, Juli 2010, S. 837–845, doi:10.1016/j.biochi.2010.03.004, PMID 20353808 (englisch).
- ↑ T. P. Soares da Costa, J. B. Christensen, S. Desbois, S. E. Gordon, R. Gupta, C. J. Hogan, T. G. Nelson, M. T. Downton, C. K. Gardhi, B. M. Abbott, J. Wagner, S. Panjikar, M. A. Perugini: Methods in Enzymology - Analytical Ultracentrifugation. Band 562, 2015, ISBN 978-0-12-802908-4, Quaternary Structure Analyses of an Essential Oligomeric Enzyme, S. 205–223, doi:10.1016/bs.mie.2015.06.020, PMID 26412653 (englisch).
- ↑ A. C. Muscroft-Taylor, T. P. Soares da Costa, J. A. Gerrard: New insights into the mechanism of dihydrodipicolinate synthase using isothermal titration calorimetry. In: Biochimie. Band 92, Nr. 3, März 2010, S. 254–262, doi:10.1016/j.biochi.2009.12.004, PMID 20025926 (englisch).
- ↑ J. B. Christensen, T. P. Soares da Costa, P. Faou, F. G. Pearce, S. Panjikar, M. A. Perugini: Structure and Function of Cyanobacterial DHDPS and DHDPR. In: Scientific Reports. Band 6, Nr. 1, November 2016, S. 37111, doi:10.1038/srep37111, PMID 27845445, PMC 5109050 (freier Volltext), bibcode:2016NatSR...637111C (englisch).
- ↑ Biolys®—the lysine source with added extras
- ↑ S. Ebel, H. J. Roth (Hrsg.): Lexikon der Pharmazie. Georg Thieme Verlag, 1987, ISBN 3-13-672201-9, S. 406.
Auf dieser Seite verwendete Medien
Autor/Urheber: Tatiana soaresdacosta, Lizenz: CC BY-SA 4.0
Lysine Biosynthesis Pathways
Autor/Urheber: Furfur, Lizenz: CC BY-SA 4.0
Methylierung der Aminosäure Lysin.
Struktur von L-Lysin
Betain-Lysin
D-Lysin





