G-Protein-gekoppelte Rezeptoren

G-Protein-gekoppelte Rezeptoren (englisch G protein-coupled receptor, GPCR) sind biologische Rezeptoren in der Zellmembran und der Membran von Endosomen, die Signale über GTP-bindende Proteine (kurz G-Proteine) in das Zellinnere beziehungsweise das Innere des Endosoms weiterleiten (Signaltransduktion). In der Neurobiologie wird für G-Protein-gekoppelte Rezeptoren häufig der Begriff metabotrope Rezeptoren verwendet, um sie von einem anderen Rezeptortyp, den ligandengesteuerten Ionenkanälen (Ionotroper Rezeptor), zu unterscheiden.[1] Diese Unterscheidung ist jedoch nicht ausreichend trennscharf, denn auch enzym-gekoppelte Rezeptoren gehören zu den metabotropen Rezeptoren.
Die Familie der G-Protein-gekoppelten Rezeptoren stellt mit mehr als 1000 verschiedenen Mitgliedern die größte Proteinsuperfamilie dar. G-Protein-gekoppelte Rezeptoren sind für die Verarbeitung von Licht-, Geruchs- und einer Vielzahl von Geschmacksreizen verantwortlich. Sie spielen eine entscheidende Rolle bei Entzündungsprozessen, der gezielten Zellbewegung (Taxis), dem Transport von Stoffen durch die Zellmembran (Endozytose und Exozytose) sowie beim Zellwachstum und bei der Zelldifferenzierung. Sie sind darüber hinaus als Zielstrukturen für die Wirkung von Hormonen, wie Adrenalin oder Glucagon, und Neurotransmittern, wie Serotonin und Acetylcholin, verantwortlich. Auch einige Viren nutzen G-Protein-gekoppelte Rezeptoren als Bindungsstellen für den Eintritt in die Zelle (beispielsweise HIV).
Definition
Als G-Protein-gekoppelte Rezeptoren im engeren Sinn werden alle heptahelikalen, das heißt mit 7 Helices in der Zellmembran verankerten Rezeptoren bezeichnet, die zur Bindung und Aktivierung von G-Proteinen befähigt sind. Zusätzlich zur Bindung von G-Proteinen sind viele dieser Rezeptoren in der Lage, auch mit anderen signalweiterleitenden Proteinen zu interagieren. Als G-Protein-gekoppelte Rezeptoren werden, wie im Fall vieler sogenannter „Orphan-GPCRs“, auch Rezeptorproteine bezeichnet, für die eine Kopplung mit G-Proteinen lediglich vermutet wird. Bisweilen wird dieser Begriff auch auf Rezeptoren angewandt, die zwar nicht zur Bindung und Aktivierung von G-Proteinen befähigt sind, jedoch auf Grund ihrer phylogenetischen Verwandtschaft mit anderen, der klassischen Definition genügenden G-Protein-gekoppelten Rezeptoren dieser Familie zugerechnet werden können. Dem gegenüber werden prokaryotische Rhodopsine, die zwar in ihrer Struktur eukaryotischen G-Protein-gekoppelten Rezeptoren ähneln, aber nicht zur Bindung von G-Proteinen befähigt sind und als Ionenkanäle fungieren, nicht der Familie der G-Protein-gekoppelten Rezeptoren zugeordnet.
Vorkommen
G-Protein-gekoppelte Rezeptoren kommen in fast allen Lebewesen vor. Besonders zahlreich sind sie bei Wirbeltieren zu finden. Aber auch Wirbellose, Protozoen (z. B. Amöben) und Pilze (beispielsweise in Hefen) besitzen G-Protein-gekoppelte Rezeptoren. Auch einige pflanzliche Zellmembranproteine können auf Grund ihrer Fähigkeit, heterotrimäre G-Proteine zu aktivieren, den G-Protein-gekoppelten Rezeptoren zugerechnet werden.[2][3] Sie spielen insbesondere eine Rolle als Phytohormon-Rezeptoren.[4] Allerdings sind nicht alle dieser pflanzlichen Rezeptoren wie der GCR1 der Acker-Schmalwand klassische heptahelikale G-Protein-gekoppelte Rezeptoren, sondern können wie GCR2, der größere Ähnlichkeiten zu bakteriellen Lanthionin-Synthasen besitzt,[5] abweichende Strukturmotive besitzen. Einige Fotorezeptoren mit einer Struktur, die G-Protein-gekoppelten Rezeptoren ähnelt, können sogar in Archaeen gefunden werden (Bacteriorhodopsine). Diese als Ionenkanäle fungierenden Rezeptoren haben jedoch keine Verwandtschaft zu Fotorezeptoren höherer Tiere und werden in der Regel nicht der Familie der G-Protein-gekoppelten Rezeptoren zugerechnet.
Beim Menschen konnten bisher etwa 800 G-Protein-gekoppelte Rezeptoren identifiziert werden. Diese werden durch etwa 3 % des menschlichen Genoms kodiert. Mehr als die Hälfte der G-Protein-gekoppelten Rezeptoren des Menschen werden den Geruchsrezeptoren (olfaktorischen Rezeptoren) zugeordnet. Bei über 140 der ca. 800 G-Protein-gekoppelten Rezeptoren ist der endogene Ligand nicht bekannt und sie werden deshalb als Orphan-GPCRs bezeichnet.[6]
Struktur

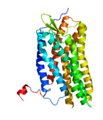
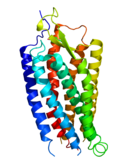
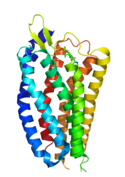
Auf Grund ihrer Struktur gehören mit Ausnahme des pflanzlichen GCR2 alle G-Protein-gekoppelte Rezeptoren der Superfamilie der heptahelikalen Transmembranproteine (gebräuchliche Synonyme: Sieben-Transmembrandomänen-Rezeptoren, 7-TM-Rezeptoren und heptahelikale Rezeptoren) an. Sie bestehen aus Untereinheiten mit sieben (griechisch „hepta“) die Zellmembran durchspannenden (transmembranären) Helixstrukturen, die durch drei intrazelluläre und drei extrazelluläre Schleifen miteinander verbunden sind. G-Protein-gekoppelte Rezeptoren besitzen eine extrazelluläre oder transmembranäre Bindungsdomäne für einen Liganden. Das G-Protein bindet an der zellinneren (intrazellulären) Seite des Rezeptors. Zum Vergleich des Aufbaus der verschiedenen G-Protein-gekoppelten Rezeptoren untereinander wurde das Ballesteros-Weinstein-System der Nomenklatur entwickelt.
Lange Zeit konnte die Struktur G-Protein-gekoppelter Rezeptoren nur anhand der Analogie zur bekannten Struktur des Bakteriorhodopsins vorausgesagt werden. Die dreidimensionale Strukturaufklärung eines G-Protein-gekoppelten Rezeptors bei Wirbeltieren, des Rhodopsins des Hausrinds, gelang im Jahr 2000 mit Hilfe der Röntgenstrukturanalyse.[7] Die Kristallisierung und Strukturaufklärung anderer G-Protein-gekoppelter Rezeptoren ist hingegen wegen ihrer physikochemischen Eigenschaften und aufgrund der geringen Rezeptordichte in der Membran erschwert. Daher konnte erst im Jahr 2007 die Kristallstruktur eines ligandenaktivierten G-Protein-gekoppelten Rezeptors (menschlicher β2-Adrenozeptor) unter Verwendung technischer Tricks, wie der Verwendung stabilisierender Antikörper oder der Fusion mit leicht zu kristallisierenden Proteinen, aufgeklärt werden.[8][9]
Inzwischen wurde die dreidimensionale Struktur, einschließlich die der Transmembrandomänen, für zahlreiche G-Protein-gekoppelte Rezeptoren der physiologisch und therapeutisch bedeutsamen Klasse A aufgeklärt. Die Strukturaufklärung erfolgte überwiegend mit Hilfe der Röntgenkristallanalyse unter Verwendung von Fusionsproteinen oder Methoden zur thermischen Stabilisierung. Auf diese Weise wurde unter anderem die Struktur des β1-Adrenozeptors,[10] des A2A-Adenosinrezeptors,[11] des D3-Dopaminrezeptors,[12] der Opioidrezeptoren κ,[13] μ,[14] δ,[15] Nociceptin,[16] des S1P1-Rezeptors,[17] der muskarinischen Acetylcholinrezeptoren M2[18] und M3,[19] des Histamin-H1-Rezeptors,[20] der Serotonin-Rezeptoren 5-HT1B und 5-HT2B,[21] der Chemokinrezeptoren CCR5[22] und CXCR4,[23] des Neurotensinrezeptors NTS1[24] und des Protease-aktivierten Rezeptors PAR1 aufgeklärt.[25] Mittels Kernspinresonanzspektroskopie wurde die dreidimensionale Struktur des natürlichen humanen Chemokinrezeptors CXCR1 ermittelt.[26] Genutzt werden die Flüssig- und Festphasenspektroskopie.[27]
Für G-Protein-gekoppelte Rezeptoren der Klasse A liegen Strukturdaten der Rezeptoren in Komplex mit Signaltransduktionsmolekülen, wie beispielsweise heterotrimäre G-Proteine und Arrestin, und für verschiedene Aktivierungszustände vor. Diese Daten liefern einen wichtigen Einblick in den molekularen Mechanismus der Rezeptoraktivierung und der Signalweiterleitung.
Von den G-Protein-gekoppelten Rezeptoren der Klasse B liegen die Strukturdaten für den Corticotropin-Releasing-Hormon-Rezeptor 1[28] und den Glucagonrezeptor vor.[29] Von den Rhodopsin-ähnlichen G-Protein-gekoppelten-Rezeptoren der Klasse A unterscheidet sich der bisher einzige strukturaufgeklärte G-Protein-Rezeptor der Klasse F, der Smoothened-Rezeptor, durch ein Fehlen vieler typischer Strukturmotive und durch ausgeprägte extrazelluläre Schleifen.[30] Für zahlreiche weitere G-Protein-gekoppelte Rezeptoren konnten zumindest Teile der Struktur, wie beispielsweise die der extrazellulären Domänen, experimentell bestimmt werden.
Auch die Sonderrolle des pflanzlichen Phytohormonrezeptors GCR2 unter den G-Protein-gekoppelten Rezeptoren konnte mit Hilfe der Röntgenstrukturanalyse bestätigt werden. Zwar wurde er auf Grund biochemischer Voraussagen und der Aktivierung durch das Phytohormon Abscisinsäure der Gruppe der G-Protein-gekoppelten Rezeptoren zugeordnet, besitzt aber wesentlich abweichende physikochemische Eigenschaften.[3][31] Mit seinen identifizierten 14 Helices ist er der einzige bekannte nicht heptahelikale G-Protein-gekoppelte Rezeptor und ähnelt strukturell eher bakteriellen Lanthionin-Synthasen.[5]
- Rhodopsin
- β1-Adrenozeptor
- β2-Adrenozeptor
- Histamin-H1-Rezeptor
- M2-Acetylcholinrezeptor
- M3-Acetylcholinrezeptor
- A2A-Adenosinrezeptor
- S1P1-Rezeptor
- CXCR1
- CXCR4
- PAR1
- Neurotensin-NTS1-Rezeptor
Transmembrandomänen

Die sieben membrandurchspannenden helikalen Domänen G-Protein-gekoppelter Rezeptoren, die bei Sicht auf den Rezeptor von der extrazellulären Seite aus entgegen dem Uhrzeigersinn angeordnet sind, betten sich aufgrund ihrer überwiegend lipophilen Außenoberfläche in eine Lipidmembran wie die Zellmembran ein. Das obere Drittel des Rezeptors beherbergt über den Kontakt zu den Transmembrandomänen III, V und VI die Bindungsstelle für einen primären (orthosterischen) Liganden, mit der Ausnahme, dass dieselbe Bindungsstelle in GPCR der Klasse C (GPCR-C) eine allosterische ist.
Im Gegensatz zu den extrazellulären und intrazellulären Domänen sind die Transmembrandomänen innerhalb der Familie G-Protein-gekoppelter Rezeptoren stark konserviert. Einige Aminosäuresequenzen (Motive) innerhalb der Transmembrandomänen sind für viele G-Protein-gekoppelte Rezeptoren charakteristisch. Beispielsweise können das E/DRY-Motiv der Transmembrandomäne III und das NPxxY-Motiv der Transmembrandomäne VII in fast allen Rhodopsin-ähnlichen Rezeptoren gefunden werden. Ihnen wird eine wichtige Rolle bei der Rezeptoraktivierung zugeschrieben.
Am Beispiel des MOR ist die Modulation eines GPCR durch Ionen seit 1973 bekannt. Die im Kern von 70 bis 90 % der GPCR-A befindliche Natriumbindungsstelle ist hoch konserviert und weit überwiegend verankert über die saure Aminosäure Aspartat in der Position D2.50. In dieser Bindungsstelle stabilisiert ein einzelnes Natriumkation antagonistische Rezeptorzustände, vermag andererseits aber auch die Efficacy von Vollagonisten zu erhöhen. Das Ion wird durch Aktivierung verdrängt, wobei die Bindungsstelle kollabiert. Andere Alkalikationen erreichen nicht die modulatorische Wirkstärke des Natriums. Der Natriumeffekt ist in manchen Rezeptortypen stark (MOR), in anderen schwach ausgeprägt (DRD4), oder er fehlt. Zahlreiche Beispiele von GPCR-A mit abweichender supramolekularer Verankerung des Natriumions, mit verminderter oder fehlender Natriumbindung sind gut untersucht. Eine identische Bindungsstelle ist in anderen GPCR-Klassen nicht identifiziert worden.[32]
Extrazelluläre Domänen

Einige G-Protein-gekoppelte Rezeptoren, wie z. B. die metabotropen Glutamatrezeptoren, besitzen in ihrer extrazellulären N-terminalen Domäne ihre primäre Ligandenbindungsstelle. Diese Rezeptoren sind durch lange N-terminale Aminosäuresequenzen gekennzeichnet (bis 2800 Aminosäuren), während Rezeptoren mit intrazellulären Ligandenbindungsdomänen meist nur kurze Reste aufweisen (meist unter 30 Aminosäuren). Der N-Terminus und die extrazellulären Domänen des Rezeptors sind häufig glykosyliert.
Große strukturelle Unterschiede konnten auch in der zweiten extrazellulären Schleife (EL 2), welche in der Nähe der Ligandenbindungsstelle der Rhodopsin-ähnlichen G-Protein-gekoppelten liegt, gefunden werden. Häufig sind entweder β-Haarnadel- oder α-Helixmotive in dieser Schleife zu finden. In der zweiten extrazellulären Schleife und am Anfang der dritten transmembranären Domäne des Rezeptors befinden sich zwei konservierte, zur Disulfidbrückenbildung befähigte Cysteine, welche die Struktur des Rezeptors stabilisieren, indem sie die Transmembrandomänen III bis V aneinanderbinden. Abweichend davon ist beispielsweise die extrazelluläre Schleife 2 des κ-Opioidrezeptors nur über eine Disulfidbrücke stabilisiert.[13]
Intrazelluläre Domänen
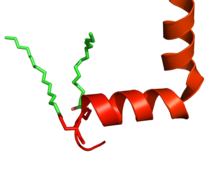
Die intrazelluläre Seite des Rezeptors ist mit Bindungsstellen für G-Proteine und andere Signalmoleküle ausgestattet. An der Bindung von G-Proteinen sind insbesondere die transmembrandomänennahen Aminosäuren der zweiten (IL 2) und dritten intrazellulären Schleife (IL 3) sowie der sich an die 7. transmembranäre Domäne anschließende C-terminale Rest beteiligt. Der intrazelluläre C-terminale Anteil ist in der Regel sehr kurz (meist unter 50 Aminosäuren). Einigen G-Protein-gekoppelten Rezeptoren, wie beispielsweise dem Gonadotropin-Releasing-Hormon-Rezeptor, fehlt dieser Teil. Direkt an das intrazelluläre Ende der 7. transmembranären Domäne kann sich auch noch eine mit einem konservierten Cystein beginnende achte Helix (Hx 8) anschließen, die parallel zur Zellmembran verläuft. Die Helix 8 trägt oft Fettsäurereste, wie Palmitinsäure- und Ölsäure-Reste, die der Verankerung dieser Helix in der Zellmembran dienen.
Funktion
Die Hauptfunktion der G-Protein-gekoppelten Rezeptoren besteht in der Weiterleitung von Signalen in das Zellinnere. Diese Signalweiterleitung (Signaltransduktion) geschieht insbesondere über die Aktivierung von G-Proteinen. Einige G-Protein-gekoppelte Rezeptoren sind auch zu einer G-Protein-unabhängigen Signaltransduktion, beispielsweise über Arrestine, befähigt.
Aktivierung von G-Proteinen
Nahezu alle G-Protein-gekoppelten Rezeptoren sind zu einer direkten Aktivierung eines aus drei Untereinheiten (α, β und γ) bestehenden (heterotrimeren) G-Proteins befähigt. Die Aktivierung eines G-Proteins ist ein mehrstufiger Prozess, der die Bindung eines Liganden an den Rezeptor, die Konformationsänderung des Rezeptors sowie die Bindung und Aktivierung eines G-Proteins einschließt und dabei den Gesetzen der Thermodynamik unterliegt.

(1) Bindung des G-Proteins; (2) Ligandenbindung; (3) Aktivierung des Rezeptors; (4) Aktivierung des G-Proteins; (5) Dissoziation des G-Proteins und Signaltransduktion; (6) Inaktivierung des G-Proteins
Schritt 1: Bindung des G-Proteins
Aktivierte Rezeptoren interagieren mit heterotrimeren G-Proteinen, die dadurch selbst aktiviert werden. Der Rezeptor zeigt dabei eine Selektivität für einen (beispielsweise β1-Adrenozeptor: Gs) oder für mehrere verschiedene (z. B. β2-Adrenozeptor: Gs und Gi/o) G-Proteinsubtypen. Dieser Komplex aus Rezeptor und heterotrimerem G-Protein ist dabei ein Bestandteil eines größeren Netzwerks, an dem auch weitere signalweiterleitende Proteine, wie z. B. GIRK-Kanäle, Phospholipase C und Proteinkinase C beteiligt sind.[33] Mit Hilfe dieses Netzwerks kann eine schnelle, oft nur Millisekunden bis wenige Sekunden dauernde Aktivierung der Signaltransduktionskaskade G-Protein-gekoppelter Rezeptoren erklärt werden. Ob der Rezeptor mit dem G-Protein durch Kollisionskopplung interagiert oder ob Rezeptoren auch im inaktiven Zustand mit dem G-Protein assoziiert sind, ist Gegenstand aktueller Forschung.[34]
Schritt 2: Ligandenbindung

Abhängig von der Art des Liganden erfolgt die Bindung an den Rezeptor an seine extrazellulären, transmembranären oder intrazellulären Domänen:
- Der als prosthetische Gruppe fungierende, kovalent gebunde Ligand 11-cis-Retinal ist fester Bestandteil des Lichtrezeptors Rhodopsin (Abb. a)[35][36] und zwar an dessen transmembranären Domänen.
- Amine (z. B. Acetylcholin, Adrenalin, Histamin und Serotonin), Nucleotide (z. B. ATP), Eikosanoide (z. B. Prostacyclin) und einige Lipide (z. B. Ceramide) binden an transmembranäre Bindungsstellen ihrer Rezeptoren (Abb. a).
- Neuropeptide (z. B. Oxytocin und Vasopressin) besetzen mehrere transmembranäre und extrazelluläre Bindungsstellen ihrer Rezeptoren gleichzeitig (Abb. b).
- Proteinasen (z. B. Thrombin und Trypsin) spalten spezifisch ein kurzes N-terminales Fragment ihrer Rezeptoren, den Protease-aktivierten Rezeptoren, ab. Durch die proteolytische Spaltung wird ein intrinsischer Ligand („tethered ligand“) am neu entstehenden Rezeptor-N-Terminus freigelegt, welcher sich in einem zweiten Schritt an eine transmembranäre Bindungsstelle anlagert (Abb. c).[37]
- Proteohormone (z. B. Glucagon) binden primär an extrazelluläre Domänen des Rezeptors. Nach einer Konformationsänderung des Rezeptors erfolgt eine sekundäre Bindung des Peptidhormons an transmembranäre Bindungsstellen (Abb. d).
- Einige kleine Stoffe (z. B. Glutamat und Calciumionen) binden ausschließlich an extrazellulären Rezeptordomänen. Durch die Anbindung des Liganden ändert sich die Konformation der extrazellulären Domänen, so dass diese mit intrazellulären Domänen in Kontakt kommen (Abb. e).
Schritt 3: Aktivierung des Rezeptors
Bindet an einen Rezeptor ein Ligand und wird dieser durch den Liganden aktiviert (Agonist), so führt diese Anlagerung meist zu einer Sprengung der Salzbrücke zwischen der 3. und der 7. transmembranären Domäne des G-Protein-gekoppelten Rezeptors. Dieser so aktivierte Rezeptor erhält mehr Flexibilität und ändert seine dreidimensionale Struktur.[38] Durch die Änderung der Konformation des Rezeptors kann dieser jetzt als GTP-Austauschfaktor (GTP exchange factor, GEF) für das gebundene G-Protein fungieren.
Viele G-Protein-gekoppelte Rezeptoren befinden sich bereits in Abwesenheit eines Agonisten in einem Gleichgewicht zwischen inaktivem und aktiviertem Zustand. Die Anbindung eines Agonisten verschiebt das Gleichgewicht in Richtung aktiver Zustand, während inverse Agonisten das Gleichgewicht in Richtung inaktiver Zustand verschieben. Rezeptoren, die sich auch in Abwesenheit eines Agonisten in einem aktivierten Zustand befinden können nennt man konstitutiv aktiv (englisch: constitutively active, abgekürzt: ca). Auch Mutationen im Rezeptor können eine konstitutive Aktivität bewirken („Constitutive active mutants“, CAM). Derartig mutierte G-Protein-gekoppelte Rezeptoren werden mit bestimmten Erkrankungen (z. B. bestimmte Formen von Retinopathia pigmentosa durch konstitutiv aktives Rhodopsin) in Verbindung gebracht und kommen in bestimmten Herpesviren vor. Weiterhin werden in der experimentellen Pharmakologie inzwischen G-Protein-gekoppelte Rezeptoren auch gezielt zu CAMs verändert, um diese bei der Suche nach neuen Medikamenten zu verwenden.
Schritt 4: Aktivierung der G-Proteine
Heterotrimere G-Proteine können GTP und GDP binden, die GDP-bindende Form ist inaktiv. Die Aktivierung des Rezeptors sorgt für den Austausch von GDP gegen GTP an der α-Untereinheit des G-Proteins. Der G-Protein-Komplex wird durch die Bindung des GTP instabil. Als Folge ändert sich die Konformation des heterotrimeren G-Proteins und es kann in die α- und die βγ-Untereinheit dissoziieren. Die aktivierten G-Proteine können nun die exogenen, durch den Liganden übertragenen Signale in das Zellinnere weiterleiten (Signaltransduktion).
Schritt 5: Signaltransduktion
Die aktivierten Untereinheiten des G-Proteins sind für die weitere Signaltransduktion verantwortlich. Je nach Untereinheit werden weitere zell- oder membranständige Proteine aktiviert oder deaktiviert. So modulieren beispielsweise die α-Untereinheiten der Gs/olf die Aktivität der Adenylylcyclase, während α-Untereinheiten der Gq/11-Proteine die Phospholipase C aktivieren. Diese Enzyme sind dann an der Bildung eines sekundären Botenstoffs beteiligt.
Schritt 6: G-Protein-Inaktivierung
Die intrinsische GTPase-Aktivität der α-Untereinheit des G-Proteins spaltet nach einer Zeit und unter Mithilfe von Proteinen, die die GTPase-Aktivität erhöhen (GTPase aktivierendes Protein, GAP), das gebundene GTP in GDP + Pi. Die α-Untereinheit des G-Proteins kann somit wieder mit der βγ-Untereinheit reassoziieren und erneut an einen Rezeptor binden (siehe Schritt 1). Es findet also eine Selbstregulierung statt.
Alternative Signaltransduktionswege
Viele G-Protein-gekoppelte Rezeptoren sind nicht nur zu einer Aktivierung von G-Proteinen, sondern auch zu einer Bindung und Aktivierung anderer, Zellprozesse steuernder Moleküle befähigt. Auf diese Weise können G-Protein-gekoppelte Rezeptoren G-Protein-unabhängig alternative Signaltransduktionswege steuern. Beispiele hierfür sind die Frizzled-Rezeptoren, die über den Wnt-Signalweg β-Catenin aktivieren können, der β2-Rezeptor, der G-Protein-unabhängig die Funktion des Na+/H+-Austauschers modulieren kann und der 5-HT2B-Rezeptor, dessen eNOS aktivierende Funktion zumindest teilweise ebenfalls G-Protein-unabhängig ist. Durch G-Protein-unabhängige Aktivierungen von Arrestin und Homer-Proteinen kann darüber hinaus die Funktion G-Protein-gekoppelter Rezeptoren selbst reguliert werden.
Regulierung der Rezeptorfunktion
Für die Funktion von G-Protein-gekoppelten Rezeptoren ist deren Lokalisation an der Zelloberfläche Voraussetzung. Zelluläre Prozesse, die zu einer Erhöhung der Rezeptorzahl an der Zellmembran und damit der Rezeptorfunktion führen, werden als Up-Regulation bezeichnet. Im Gegensatz dazu stellt die Entfernung funktionstüchtiger Rezeptoren von der Zellmembran (Down-Regulation) einen Mechanismus der Beendigung der Rezeptorfunktion dar.
Up-Regulation
Die Heraufregulierung („Up-Regulation“) G-Protein-gekoppelter Rezeptoren ist ein mehrstufiger Prozess. Ausgangspunkt ist die Proteinbiosynthese des Rezeptors am endoplasmatischen Reticulum, die unter anderem durch G-Protein-gekoppelte Rezeptoren indirekt reguliert werden kann.
Der Transport des so synthetisierten Rezeptors über Golgi-Vesikel zur Zellmembran kann durch Chaperone und Chaperon-ähnliche Proteine einschließlich Rezeptoraktivität-modifizierender Proteine gesteuert werden. Eine Regulation des Membrantransports ist ebenfalls durch eine Glykosylierung des Rezeptors möglich. Eine Bedeutung der für viele G-Protein-gekoppelte Rezeptoren beobachteten Di- und Oligomerisierung als Voraussetzung für deren Transport zur Zellmembran und somit für deren Funktionalität wird ebenfalls diskutiert. Der Einbau der G-Protein-gekoppelten Rezeptoren in die Zellmembran kann beispielsweise durch Homer-Proteine, PSD-95 und Spinophilin gesteuert werden.[39]
Down-Regulation
Die durch G-Protein-gekoppelte Rezeptoren vermittelten Effekte nehmen nach längerer Zeit der Aktivierung ab. Eine Schlüsselrolle dieser Herabregulierung („Down-Regulation“) spielt dabei die Phosphorylierung intrazellulärer Domänen des Rezeptors (C-terminale Serin- oder Threonin-Reste) durch Proteinkinasen.
Phosphorylierung durch Second-Messenger-aktivierte Kinasen
Proteinkinasen, die über G-Protein-vermittelte Produktion von Second Messengern (sekundären Botenstoffen) aktiviert werden (wie beispielsweise die durch cyclisches Adenosinmonophosphat (cAMP) aktivierte Proteinkinase A oder die durch Diacylglycerol und Calcium aktivierte Proteinkinase C), können viele G-Protein-gekoppelte Rezeptoren phosphorylieren. Häufig wird dadurch die Signaltransduktion über den Rezeptor unterbrochen, da der phosphorylierte Rezeptor eine geringere Affinität zu G-Proteinen besitzt. Diese Regulation kann demzufolge als ein negativer Rückkopplungs-Mechanismus wirken.
Phosphorylierung durch G-Protein-gekoppelte Rezeptorkinasen
G-Protein-gekoppelte Rezeptorkinasen (kurz GRKs) haben im Wesentlichen zwei Funktionen. Erstens können sie mit den Gα- und Gβγ-Untereinheiten der heterotrimeren G-Proteine interagieren, womit diese nicht mehr zur Signaltransduktion beitragen. Außerdem können sie als GAP (GTPase-aktivierendes Protein) wirken und die Umwandlung von GTP*Gα zu GDP*Gα beschleunigen. Zweitens sind sie Serin-/Threonin-Kinasen und können als solche G-Protein-gekoppelte Rezeptoren phosphorylieren.
Folgen der Phosphorylierung
- Konformationsänderung des Rezeptors: durch die stark negative Ladung des Phosphatrests und damit einhergehende elektrostatische Wechselwirkungskräfte ändert sich die Konformation des Rezeptors. Die neue Konformation ist oft ungünstiger für die Rezeptor-G-Protein-Interaktion oder beeinflusst die Affinität des Rezeptors, so dass es zu einer Abschwächung des Rezeptorsignals kommt. Diese Art der Desensitivierung erfolgt oft durch die Second-Messenger-abhängigen Proteinkinasen A und C.
- Interaktion mit beta-Arrestinen: Durch Bindung von Arrestin an den vor allem durch GRKs phosphorylierten Rezeptor wird sterisch eine Anbindung der G-Proteine verhindert (= Kurzzeitregulation innerhalb weniger Minuten). Außerdem dienen die beta-Arrestine als „scaffold“-Moleküle für eine Vielzahl weiterer Proteine, besonders hervorzuheben sind dabei Clathrin und die MAP-Kinasen.
- Internalisierung: Die Bindung des Rezeptor-Arrestin-Komplexes an Clathrin führt zur Entfernung des phosphorylierten Rezeptors von der Zelloberfläche ins Zellinnere in Form von Membranvesikeln (clathrin-coated pits). Nachfolgend kann der Rezeptor intrazellulär abgebaut, recycelt und damit an die Oberfläche zurückgebracht werden oder auch als intrazellulärer Rezeptor fortbestehen. Diese Regulation erfolgt meist innerhalb von 10 bis 30 Minuten und wird nach Entfernung des Rezeptorstimulus oft innerhalb von 30 bis 60 Minuten rückgängig gemacht. Eine langfristige Regulation über Tage oder Monate ist oft auf Transkriptionsregulation zurückzuführen.
Die Bindung von MAP-Kinasen an den Rezeptor-Arrestin-Komplex führt dazu, dass diese nicht mehr über G-Proteine, sondern direkt vom Rezeptor stimuliert werden. Dieser Wechsel des Signaltransduktionsmechanismus erfolgt ebenfalls innerhalb von etwa zehn Minuten.
Einteilung
Klassifizierung nach Funktion

Eine erste systematische Klassifizierung der G-Protein-gekoppelten Rezeptoren erfolgte Anfang der 1990er Jahre anhand funktioneller Merkmale und wird bis heute weitergeführt:
- Klasse A (oder 1) (Rhodopsin-ähnlich (Rhodopsin))
- Klasse B (oder 2) (Sekretin-Rezeptor-Familie (Sekretin))
- Klasse C (oder 3) (metabotropen Glutamat/Pheromon (Glutamatrezeptor))
- Klasse D (oder 4) (Fungal mating pheromone receptors)
- Klasse E (oder 5) (cyclische AMP-Rezeptoren (cAMP))
- Klasse F (oder 6) (Frizzled/Smoothened (Wnt-Signalweg))
Anhand dieses Systems wurden die G-Protein-gekoppelten Rezeptoren von Wirbeltieren und Wirbellosen in fünf Gruppen (A–E) unterteilt. Die Gruppe A repräsentierten mit Rhodopsin verwandte Rezeptoren, Glycoproteinrezeptoren wurden in die Gruppe B und die metabotropen Glutamatrezeptoren in die Gruppe C eingeteilt. Rezeptoren der Gruppen D und E kommen nicht bei Wirbeltieren vor. Sie fungieren als Pheromonrezeptoren in Hefen bzw. als cAMP-Rezeptoren in Nematoden. Mit der Entdeckung neuer G-Protein-gekoppelter Rezeptoren wurde dieses System in den letzten Jahren erweitert. Außerhalb des oben beschriebenen ABCDE-Systems wurden eigene Gruppen für die G-Protein-gekoppelten-Rezeptoren der Pflanzen, für die Geruchsrezeptoren der Insekten, für die Chemorezeptoren der Nematoden und für die „Frizzeled/Smoothened“-Rezeptoren höherer Tiere etabliert.[40]
Phylogenetische Klassifikation
Ein alternatives System der Klassifizierung der humanen G-Protein-gekoppelten Rezeptoren wurde basierend auf phylogenetischen Untersuchungen vorgeschlagen (GRAFS- oder Fredriksson-System). Diesem System zufolge werden G-Protein-gekoppelte Rezeptoren in fünf Hauptgruppen unterteilt: in die Glutamat-, Rhodopsin-, Adhäsions-, Frizzled/Taste2- und Secretin-Gruppe.[41]
Bedeutung der G-Protein-gekoppelten Rezeptoren für die Medizin
Arzneistoffe
In der modernen Medizin nehmen G-Protein-gekoppelte Rezeptoren eine Schlüsselposition ein: Etwa 30 %[42] aller verschreibungspflichtigen Medikamente, die derzeit auf dem Markt sind, wirken auf G-Protein-gekoppelte Rezeptoren ein. Unter diesen Medikamenten befinden sich unter anderem die häufig verschriebenen Betablocker, Neuroleptika, Antihistaminika, Opioide und Sympathomimetika. Neue, über G-Protein-gekoppelte Rezeptoren wirkende Medikamente, wie beispielsweise die Triptane, Setrone und Sartane, haben in den letzten Jahren ebenfalls einen hohen Stellenwert erreicht.
| Arzneistoffgruppe | Beispiele | Indikation | Rezeptor(en) | Erläuterungen |
|---|---|---|---|---|
| α2-Agonisten | Clonidin | Arterielle Hypertonie | α2-Adrenozeptoren | Senken als Agonisten an α2-Adrenozeptoren im Zentralnervensystem die Aktivität des Sympathikus und reduzieren darüber den Blutdruck. |
| Alphablocker | Prazosin, Tamsulosin | Arterielle Hypertonie, Prostatahyperplasie | α1-Adrenozeptoren | Senken als Antagonisten an α1-Adrenozeptoren den Tonus der glatten Muskulatur in Blutgefäßen und im Urogenitaltrakt. |
| Anticholinergika | Atropin, Ipratropium, Tiotropium | Harninkontinenz, Asthma bronchiale, bradykarde Herzrhythmusstörungen | Muscarinische Acetylcholinrezeptoren | Senken als Antagonisten an Muskarin-Rezeptoren den Tonus der glatten Muskulatur in Bronchien und im Urogenitaltrakt. Hemmen die Herzfrequenz-senkende Wirkung von Acetylcholin. |
| H1-Antihistaminika | Diphenhydramin, Loratadin, Cetirizin | Allergische Reaktionen | H1-Rezeptoren | Hemmen als Antagonisten die Wirkung von Histamin, das bei allergischen Reaktionen ausgeschüttet wird, an H1-Rezeptoren. |
| H2-Antihistaminika | Ranitidin, Famotidin, Cimetidin | Kontrolle der Magensäureproduktion (Refluxkrankheiten, Magengeschwür) | H2-Rezeptoren | Hemmen als Antagonisten an H2-Rezeptoren die Histamin-vermittelte Freisetzung von Magensäure. |
| AT1-Antagonisten | Losartan, Candesartan, Irbesartan, Valsartan, Telmisartan | Hypertonie, Herzinsuffizienz, koronare Herzkrankheit | AT1-Rezeptoren | Senken als Antagonisten des Angiotensins II an AT1-Rezeptoren den Tonus der glatten Muskulatur in Blutgefäßen. |
| Betablocker | Atenolol, Bisoprolol, Carvedilol, Metoprolol, Timolol | Arterielle Hypertonie, Herzinsuffizienz, koronare Herzkrankheit, Migräne, Glaukom | β1-Adrenozeptoren | Senken als Antagonisten an β1-Adrenozeptoren unter anderem die Herzfrequenz. Senkung des Augeninnendrucks. |
| Bradykininrezeptorantagonisten | Icatibant | Hereditäres Angioödem | Bradykininrezeptoren | Blockieren als Antagonisten die Entzündungssymptome vermittelnden Wirkungen des Bradykinins. |
| Dopamin-Agonisten | Pergolid, Cabergolin, Pramipexol, Ropinirol | Parkinson-Krankheit, Restless-Legs-Syndrom | Dopamin-Rezeptoren | Imitieren als Agonisten an Dopaminrezeptoren die Wirkung des Dopamins. |
| Endothelin-Rezeptorantagonisten | Bosentan, Sitaxentan, Ambrisentan | Pulmonale Hypertonie | Endothelin-Rezeptoren | Senken als Antagonisten an ET1-Rezeptoren den Tonus und das Remodelling der Lungenblutgefäße. |
| Korezeptor-Antagonisten | Maraviroc | HIV | Chemokinrezeptoren CCR5 oder CXCR4 | Blockieren die für den Eintritt von HI-Viren nötigen Chemokinrezeptoren. |
| NK1-Rezeptorantagonisten | Aprepitant | Erbrechen | NK1-Rezeptoren | Hemmen als Antagonisten NK1-Rezeptoren im Brechzentrum. |
| Neuroleptika | Haloperidol, Risperidon, Clozapin, Olanzapin | Schizophrenie | D2-Rezeptoren, 5-HT2A-Rezeptoren | Führen über eine vorrangige Hemmung von D2-Rezeptoren (typische Neuroleptika) oder 5-HT2A-Rezeptoren (atypische Neuroleptika) zu einer antipsychotischen Wirkung. |
| Opioide | Morphin, Codein, Fentanyl, Loperamid | Schmerzen, Anästhesie, Husten, Durchfall | Opioidrezeptoren | Führen als Agonisten an den Opioidrezeptoren μ,κ und δ zu einer spinalen und supraspinalen Analgesie sowie zu einer zentralen Hemmung des Hustenreizes. |
| S1P-Rezeptormodulatoren | Fingolimod | Multiple Sklerose | Sphingosin-1-phosphat-Rezeptoren | Internalisierung von S1P-Rezeptoren. |
| Triptane | Sumatriptan, Naratriptan, Zolmitriptan | Migräne | 5-HT1B-Rezeptoren | Migränetherapeutische Wirkung über eine Stimulation von 5-HT1B-Rezeptoren in zerebralen Blutgefäßen und Neuronen. |
Forschung

Das Prinzip der Signalübertragung von Hormonen mittels Rezeptoren und sekundärer Botenstoffe wurde 1960 erstmals postuliert, die Übertragung durch G-Proteine wurde in den 1970er bis 1980er Jahren erarbeitet. Für diese Arbeiten wurden 1971 Earl W. Sutherland Jr. „für seine Entdeckungen über die Wirkungsmechanismen von Hormonen“ und 1994 Alfred G. Gilman und Martin Rodbell „für die Entdeckung der Zellkommunikation und im Speziellen der Entdeckung der G-Proteine“ mit dem Nobelpreis für Physiologie oder Medizin geehrt. 2012 wurden Brian Kobilka und Robert Lefkowitz für die Arbeiten über G-Protein-gekoppelte Rezeptoren mit dem Nobelpreis für Chemie geehrt.
G-Protein-gekoppelte Rezeptoren gehören nach wie vor zu den am intensivsten untersuchten Zielen für die Entwicklung neuer Medikamente in der Arzneimittelindustrie. Dabei rücken insbesondere neue, innerhalb der letzten 20 Jahre entdeckte Rezeptoren, wie beispielsweise Cannabinoid-Rezeptoren, CGRP-Rezeptoren, Chemokin-Rezeptoren, Endothelin-Rezeptoren, Leptin-Rezeptoren, Neurokinin-Rezeptoren und Neuropeptid-Y-Rezeptoren, in das Interesse der Forschung.
Rezeptor-Oligomere
Literatur
- T. H. Ji, M. Grossmann, I. Ji: G protein-coupled receptors. I. Diversity of receptor-ligand interactions. In: J. Biol. Chem., 273, 1998, S. 17299–17302.
- U. Gether, B. K. Kobilka: G protein-coupled receptors. II. Mechanism of agonist activation. In: J. Biol. Chem., 273, 1998, S. 17979–17982.
- R. J. Lefkowitz: G protein-coupled receptors. III. New roles for receptor kinases and beta-arrestins in receptor signaling and desensitization. In: J. Biol. Chem., 273, 1998, S. 18677–18680.
- K. L. Pierce, R. T. Premont, R. J. Lefkowitz: Seven-transmembrane receptors. In: Nat Rev Mol Cell Biol. 9, 2002, S. 639–50
- R. J. Lefkowitz, E. J. Whalen: Beta-arrestins: traffic cops of cell signaling. In: Curr. Opin. Cell Biol., 16, 2004, S. 162–168.
- R. J. Lefkowitz, S. K. Shenoy: Transduction of receptor signals by beta-arrestins. In: Science 308, 2005, S. 512–517.
- R. Fredriksson, H. B. Schiöth: The Repertoire of G-Protein–Coupled Receptors in Fully Sequenced Genomes. In: Mol Pharmacol., 67, 2005, S. 1414–1425.
- S. J. Hill: G-protein-coupled receptors: past, present and future. In: Br J Pharmacol., 147, 2006, S. S27–S37.
- Harvey Lodish, Arnold Berk, S. Lawrence Zipursky, Paul Matsudaira, David Baltimore: Molekulare Zellbiologie. 4. Auflage, S. 922 ff.
- Alberts et al.: Molecular Biology of the Cell. 4th ed., S. 852 ff.
Weblinks
- Klassifizierung der G-Protein-gekoppelten Rezeptoren (englisch)
- IUPHAR Receptor Database – ausführliche Liste GPCR-artiger Rezeptoren der International Union of Pharmacology (englisch)
- Modern Drug Discovery - It’s a GPCR world. (englisch)
- GPCRDB Tools – Visualisierung und Analyse von GPCR Daten (englisch)
Einzelnachweise
- ↑ R. Irannejad, J. C. Tomshine u. a.: Conformational biosensors reveal GPCR signalling from endosomes. In: Nature. Band 495, Nummer 7442, März 2013, S. 534–538, doi:10.1038/nature12000, PMID 23515162.
- ↑ S. Pandey, S. M. Assmann: The Arabidopsis putative G protein-coupled receptor GCR1 interacts with the G protein alpha subunit GPA1 and regulates abscisic acid signaling. In: Plant Cell. 16. Jahrgang, 2004, S. 1616–1632.
- ↑ a b X. Liu, Y. Yue, B. Li et al.: A G protein-coupled receptor is a plasma membrane receptor for the plant hormone abscisic acid. In: Science. 315. Jahrgang, Nr. 5819, März 2007, S. 1712–1716, doi:10.1126/science.1135882, PMID 17347412.
- ↑ N. Tuteja: Signaling through G protein coupled receptors. In: Plant Signal Behav. 4. Jahrgang, Nr. 10, Oktober 2009, S. 942–947, PMID 19826234, PMC 2801357 (freier Volltext).
- ↑ a b J.-H. Chen, J. Guo, J.-G. Chen, S. K. Nair: Crystal Structure of Arabidopsis GCR2. 2013, abgerufen am 7. Mai 2013 (10.2210/pdb3t33/pdb).
- ↑ A. Levoye, J. Dam, M. A. Ayoub, J. L. Guillaume, R. Jockers: Do orphan G-protein-coupled receptors have ligand-independent functions? New insights from receptor heterodimers. In: EMBO reports. Band 7, Nummer 11, November 2006, S. 1094–1098, doi:10.1038/sj.embor.7400838, PMID 17077864, PMC 1679777 (freier Volltext).
- ↑ K. Palczewski, T. Kumasaka, T. Hori, C. A. Behnke, H. Motoshima, B. A. Fox, I. Le Trong, D. C. Teller, T. Okada, R. E. Stenkamp, M. Yamamoto, M. Miyano: Crystal structure of rhodopsin: A G protein-coupled receptor. In: Science. Band 289, Nummer 5480, August 2000, S. 739–745, PMID 10926528.
- ↑ S. G. Rasmussen et al.: Crystal structure of the human β2 adrenergic G-protein-coupled receptor. In: Nature. 450. Jahrgang, Nr. 7168, 2007, S. 383–387, PMID 17952055.
- ↑ V. Cherezov et al.: High-resolution crystal structure of an engineered human beta2-adrenergic G protein-coupled receptor. In: Science. 318. Jahrgang, Nr. 5854, 2007, S. 1258–1265, PMID 17962520.
- ↑ T. Warne et al.: Structure of a beta(1)-adrenergic G-protein-coupled receptor. Nature. 2008 Jun 25. Epub ahead of print, PMID 18594507
- ↑ V Jaakola et al.: The 2.6Å Crystal Structure of a Human A2A Adenosine Receptor Bound to an Antagonist. In: Science, Epub 2008 Oct 2, PMID 18832607
- ↑ Protein Data Bank: 3PBL Structure Summary
- ↑ a b H. Wu et al.: Structure of the human κ-opioid receptor in complex with JDTic. In: Nature. 485. Jahrgang, Nr. 7398, 2012, S. 327–332, doi:10.1038/nature10939, PMID 22437504.
- ↑ A Manglik et al. (2012) PMID 22437502
- ↑ S. Granier et al. (2012) PMID 22596164
- ↑ A. A. Thompson, W. Liu, E. Chun, V. Katritch, H. Wu, E. Vardy, X. P. Huang, C. Trapella, R. Guerrini, G. Calo, B. L. Roth, V. Cherezov, R. C. Stevens: Structure of the nociceptin/orphanin FQ receptor in complex with a peptide mimetic. In: Nature. Band 485, Nummer 7398, Mai 2012, S. 395–399, doi:10.1038/nature11085, PMID 22596163, PMC 3356928 (freier Volltext).
- ↑ M. A. Hanson et al.: Crystal structure of a lipid G protein-coupled receptor. In: Science. 335. Jahrgang, Nr. 6070, 2012, S. 851–855, doi:10.1126/science.1215904, PMID 22344443.
- ↑ K. Haga et al.: Structure of the human M2 muscarinic acetylcholine receptor bound to an antagonist. In: Nature. 482. Jahrgang, Nr. 7386, 2012, S. 547–551, doi:10.1038/nature10753, PMID 22278061.
- ↑ A. C. Kruse, J. Hu, A. C. Pan, D. H. Arlow, D. M. Rosenbaum, E. Rosemond, H. F. Green, T. Liu, P. S. Chae, R. O. Dror, D. E. Shaw, W. I. Weis, J. Wess, B. K. Kobilka: Structure and dynamics of the M3 muscarinic acetylcholine receptor. In: Nature. Band 482, Nummer 7386, Februar 2012, S. 552–556, doi:10.1038/nature10867, PMID 22358844, PMC 3529910 (freier Volltext).
- ↑ T. Shimamura et al.: Structure of the human histamine H1 receptor in complex with doxepin. In: Nature. 475. Jahrgang, Nr. 7354, 2012, S. 65–70, doi:10.1038/nature10236, PMID 21697825.
- ↑ C. Wang, Y. Jiang, J. Ma et al.: Structural Basis for Molecular Recognition at Serotonin Receptors. In: Science. März 2013, doi:10.1126/science.1232807, PMID 23519210.
- ↑ Q. Tan, Y. Zhu, J. Li, Z. Chen, G. W. Han, I. Kufareva, T. Li, L. Ma, G. Fenalti, J. Li, W. Zhang, X. Xie, H. Yang, H. Jiang, V. Cherezov, H. Liu, R. C. Stevens, Q. Zhao, B. Wu: Structure of the CCR5 chemokine receptor-HIV entry inhibitor maraviroc complex. In: Science. 341. Jahrgang, Nr. 6152, 2013, S. 1387–1390, doi:10.1126/science.1241475, PMID 24030490.
- ↑ B. Wu, E. Y. Chien, C. D. Mol et al.: Structures of the CXCR4 Chemokine GPCR with Small-Molecule and Cyclic Peptide Antagonists. In: Science. Oktober 2010, doi:10.1126/science.1194396, PMID 20929726.
- ↑ J. F. White et al.: Structure of the agonist-bound neurotensin receptor. In: Nature. 490. Jahrgang, Nr. 7421, 2012, S. 508–513, doi:10.1038/nature11558, PMID 23051748.
- ↑ C. Zhang et al.: High-resolution crystal structure of human protease-activated receptor 1. In: Nature. Epub ahead of print. Jahrgang, 2012, doi:10.1038/nature11701, PMID 23222541.
- ↑ S. H. Park et al.: Structure of the chemokine receptor CXCR1 in phospholipid bilayers. In: Nature. 491. Jahrgang, Nr. 7426, 2012, S. 779–783, doi:10.1038/nature11580, PMID 23086146.
- ↑ Casey O’Connor, Kate L. White, Nathalie Doncescu, Tatiana Didenko, Bryan L. Roth, Georges Czaplicki, Raymond C. Stevens, Kurt Wüthrich, Alain Milon: NMR structure and dynamics of the agonist dynorphin peptide bound to the human kappa opioid receptor. In: Proceedings of the National Academy of Sciences. 112. Jahrgang, Nr. 38, 2015, S. 11852–11857, doi:10.1073/pnas.1510117112.
- ↑ K. Hollenstein, J. Kean, A. Bortolato, R. K. Cheng, A. S. Doré, A. Jazayeri, R. M. Cooke, M. Weir, F. H. Marshall: Structure of class B GPCR corticotropin-releasing factor receptor 1. In: Nature. 499. Jahrgang, Nr. 7459, 2013, S. 438–443, doi:10.1038/nature12357, PMID 23863939.
- ↑ F. Y. Siu, M. He, C. de Graaf, G. W. Han, D. Yang, Z. Zhang, C. Zhou, Q. Xu, D. Wacker, J. S. Joseph, W. Liu, J. Lau, V. Cherezov, V. Katritch, M. W. Wang, R. C. Stevens: Structure of the human glucagon class B G-protein-coupled receptor. In: Nature. 499. Jahrgang, Nr. 7459, 2013, S. 444–449, doi:10.1038/nature12393, PMID 23863937.
- ↑ C. Wang, H. Wu, V. Katritch et al.: Structure of the human smoothened receptor bound to an antitumour agent. In: Nature. Mai 2013, doi:10.1038/nature12167, PMID 23636324.
- ↑ C. A. Johnston, B. R. Temple, J. G. Chen et al.: Comment on “A G protein coupled receptor is a plasma membrane receptor for the plant hormone abscisic acid”. In: Science. 318. Jahrgang, Nr. 5852, November 2007, S. 914; author reply 914, doi:10.1126/science.1143230, PMID 17991845.
- ↑ Zarzycka B, Zaidi SA, Roth BL, Katritch V: Harnessing Ion-Binding Sites for GPCR Pharmacology. In: Pharmacol Rev. 71. Jahrgang, Nr. 4, 2019, S. 571–595, doi:10.1124/pr.119.017863, PMID 31551350, PMC 6782022 (freier Volltext) – (englisch).
- ↑ S. Tsunoda, J. Sierralta, Y. Sun, R. Bodner, E. Suzuki, A. Becker, M. Socolich, C. S. Zuker: A multivalent PDZ-domain protein assembles signalling complexes in a G-protein-coupled cascade. In: Nature, 388, 1997, S. 243–249.
- ↑ M. Nobles, A. Benians, A. Tinker: Heterotrimeric G proteins precouple with G protein-coupled receptors in living cells. In: Proc. Natl. Acad. Sci. USA, 120, 2005, S. 18706–18711.
- ↑ Jeremy M. Berg, John L. Tymoczko, Lubert Stryer: Biochemistry. 5. Auflage. Freeman, New York 2002, ISBN 0-7167-4684-0, online verfügbar beim NCBI Bookshelf.
- ↑ Püschel et al.: Taschenlehrbuch Biochemie. Thieme-Verlag, 2011, S. 318 (online).
- ↑ C. Patterson, G. A. Stouffer, N. Madamanchi, M. S. Runge: New tricks for old dogs: nonthrombotic effects of thrombin in vessel wall biology. In: Circulation research. Band 88, Nummer 10, Mai 2001, S. 987–997, PMID 11375267 (Review).
- ↑ B. Trzaskowski, D. Latek, S. Yuan, U. Ghoshdastider, A. Debinski, S. Filipek: Action of molecular switches in GPCRs–theoretical and experimental studies. In: Current medicinal chemistry. Band 19, Nummer 8, 2012, S. 1090–1109, PMID 22300046, PMC 3343417 (freier Volltext) (Review).
- ↑ C. M. Tan, A. E. Brady, H. H. Nickols, Q. Wang, L. E. Limbird: Membrane trafficking of G protein-coupled receptors. In: Annu. Rev. Pharmacol. Toxicol., 44, 2004, S. 559–609.
- ↑ GPCRDB:Information system for G protein-coupled receptors ( vom 25. April 2019 im Internet Archive)
- ↑ R. Fredriksson, M. C. Lagerstrom, L. G. Lundin, H. B. Schiöth: The G-protein-coupled receptors in the human genome form five main families. Phylogenetic analysis, paralogon groups, and fingerprints. In: Mol. Pharmacol. 63, 2003, S. 1256–1272, PMID 12761335
- ↑ Joachim Rassow, Karin Hauser, Roland Netzker, Rainer Deutzmann: Biochemie (= Duale Reihe). 4. Auflage. Thieme, Stuttgart 2016, ISBN 978-3-13-125354-5, S. 561.
Auf dieser Seite verwendete Medien
Autor/Urheber: S. Jähnichen, Lizenz: CC BY-SA 3.0
3D Structure model ofthe double palmitoylated helix 8 of bovine rhodopsin. Data from PDB 1L9H.
Classification scheme of G protein–coupled receptor.
Class A (Rhodopsin-like),
Class B (Secretin-like),
Class C (Glutamate Receptor-like),
Autor/Urheber: S. Jähnichen, Lizenz: CC0
3D structure model of the muscarinic M2 acetylcholine receptor in complex with a ligand based on PDB 3UON
Autor/Urheber: Der ursprünglich hochladende Benutzer war Sven Jähnichen in der Wikipedia auf Deutsch, Lizenz: CC BY-SA 3.0
erstellt am: 15.05.04
erstellt von: Sven Jähnichen
Autor/Urheber: S. Jähnichen, Lizenz: CC0
3D structure model of the neurotensin receptor NTS1 in complex with NTS(8-13) based on PDB 4GRV
Autor/Urheber: Dcirovic, Lizenz: CC BY-SA 3.0
An animated view of the PDB 4DJH structure showing the human k-opioid receptor in complex with the JDTic ligand.
Autor/Urheber: Bengt Nyman, Lizenz: CC BY 2.0
Brian Kobilka and Robert Lefkowitz, Nobel Prize winners in chemistry 2012
Autor/Urheber: S. Jähnichen, Lizenz: CC0
3D structure model of the protease activated receptor PAR1 in complex with vorapaxar based on PDB 3VW7
Autor/Urheber: Sven Jähnichen, Lizenz: CC BY-SA 3.0
Activation cycle of G-proteins by G-protein-coupled receptors
3D structure model of bovine rhodopsin. Derived from the 2.6 Å crystal stucture of rhodopsin (1L9H) with covalently linked retinal and palmityl residues (grey). Structural informations were obtained from pdb.org and rendered using PyMol 0.99.
Blue: TMI.
Lightblue: TMII.
Cyan: TMIII.
Green: TMIV.
Yellow: TMV.
Organge: TMVI.
Red-orange: TMVII.
Red: Hx8.
Crystal structure of the beta-2 adrenergic receptor (ADRB2) with its inverse agonist carazolol. Derived from the 2.4 Å crystal stucture of the bata-2 adrenoceptor/T4 lysozyme fusion protein (2RH1, Cherezov V et al. (2007). High-Resolution Crystal Structure of an Engineered Human β2-Adrenergic G Protein Coupled Receptor. Science (in press). PMID: 17962520). Structural informations were obtained from pdb.org and modelled using spdbv.
Blue: TMI.
Lightblue: TMII.
Cyan: TMIII.
Green: TMIV.
Lightgreen: Helix in ECL2.
Yellow: TMVV.
Organge: TMVI.
Red-orange: TMVII.
Red: Hx8.
Autor/Urheber: S. Jähnichen, Lizenz: CC0
Structure model of the human CXCR1 receptor based on NMR data from PDB entry 2LNL
Autor/Urheber: S. Jähnichen, Lizenz: CC0
3D structure model of CXCR4 in complex with IT1t based on PDB 3ODU
Autor/Urheber: S. Jähnichen, Lizenz: CC0
3D structure modes of the muscarinic M3 acetylcholine receptor in complex with tiotropium based on PDB 4DAJ
Autor/Urheber: S. Jähnichen, Lizenz: CC0
Structure model of the ECL2 loop of the beta-2 adrenoceptor based on PDB 2RH1
Autor/Urheber: S. Jähnichen, Lizenz: CC BY-SA 3.0
3D structure of a heat stabilised mutant turkey beta-1 adrenoceptor (cartoon) bound to isoprenaline (sticks) based on crystal structure data from Warne et al. (2011). The structural basis for agonist and partial agonist action on a β(1)-adrenergic receptor. Nature 469:241-244. Structure from PDB 2Y03 rendered with PyMol
Autor/Urheber: S. Jähnichen, Lizenz: CC0
Structure model of rhodopsin based on PDB entry 1L9H showing the numbering of the transmembrane helices. Rendered using PyMol 1.4.1
Autor/Urheber: S. Jähnichen, Lizenz: CC0
3D structure model of the S1P1 receptor in complex with an antagonist based on PDB 3V2Y
Autor/Urheber: S. Jähnichen, Lizenz: CC0
3D structure model of the histamine H1 receptor in complex with doxepin based on PDB 3RZE
Autor/Urheber: S. Jähnichen, Lizenz: CC BY-SA 3.0
3D structure model of the A2A adenosine receptor bound to adenosine besed on PDB 2YDO