Mimiviridae
| Mimiviridae | ||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
 | ||||||||||||||
| Systematik | ||||||||||||||
| ||||||||||||||
| Taxonomische Merkmale | ||||||||||||||
| ||||||||||||||
| Wissenschaftlicher Name | ||||||||||||||
| Mimiviridae | ||||||||||||||
| Links | ||||||||||||||
|

Mimiviridae ist eine Familie von Riesenviren der Ordnung Imitervirales (Klasse Megaviricetes im Phylum Nucleocytoviricota).[2][A. 1] Wie bei allen Riesenviren der Nucleocytoviricota ist das Genom der Mimiviridae unsegmentiert (monopartit) und besteht aus einem linearen Doppelstrang-DNA-Molekül mit großer Länge (wie für Riesenviren üblich).
Bis zur Reorganisation der Imitervirales Anfang April 2023 durch das International Committee on Taxonomy of Viruses (ICTV) gehörten zur Familie Mimiviridae offiziell nur die beiden Gattungen Mimivirus und Rheavirus (letztere noch mit der alten Bezeichnung Cafeteriavirus) mit jeweils einer Spezies (Art), Acanthamoeba-polyphaga Mimivirus (ApMV) bzw. Cafeteria-roenbergensis Virus (CroV) an.
Mit der Reorganisation im April 2023 hat das ICTV eine Reihe als „Extended Mimiviridae“ bezeichneten Kandidaten aus der Verwandtschaft dieser beiden Spezies in neu geschaffene Familien der Imitervirales gestellt, so dass die Familie Mimiviridae die kleinste Klade (Verwandtschaftsgruppe) von Viren darstellt, zu der die beiden ursprünglichen Spezies ApMV und CroV gehören. Dabei wurde gleichzeitig die Familie Mimiviridae in Unterfamilien aufgeteilt, wobei etliche für die Gattung Mimivirus vorgeschlagene Kandidaten in neuen, eigenen Gattungen ihre Heimat fanden.[1][4][A. 2]
Systematik

Mit der Reorganisation der Ordnung Imitervirales durch das IVTC Anfang April 2023 wurden in der Familie Mimiviridae drei Unterfamilien eingerichtet, sowie weitere Gattungen wie Cotonvirus, Megavirus, Moumouvirus, Tupanvirus. Diese neuen Gattungen ersetzen auch die frühere provisorische Aufteilung der Gattung Mimivirus in Subkladen. Weitere frühere Zuordnungen von Kandidaten zur Gattung Mimivirus (wie z. B. „Mimivirus LCMiAC01“ und „02“ oder „gvSAG AB-566-O17“) sind in diesem Licht nicht mehr gültig, denn diese Vorschläge könnten jetzt in eine der neuen Gattungen zu klassifizieren sein.[4]
Die hier wiedergegebene Systematik basiert auf der Master Species List (MSL) #38 des International Committee on Taxonomy of Viruses (ICTV) vom 8. April 2023[1] und dem vorausgegangenen Vorschlag zur Reorganisation der Ordnung Imitervirales durch Aylward et al. (2021),[4] ergänzt um Vorschläge in doppelten Anführungszeichen nach der NCBI Taxonomie vom 1. Mai 2023:[10]
- Familie Mimiviridae (Mimiviriden)
- Klade – ursprünglich als Unterfamilie „Aquavirinae“ („Aquaviren“) vorgeschlagen[6][7] (nicht realisiert)
- Unterfamilie Aliimimivirinae (Mimiviridae Gruppe II, Cafeteriaviren)[11]
- Gattung Rheavirus (früher Cafeteriavirus)
- Spezies Rheavirus sinusmexicani (Cafeteria-roenbergensis-Virus, CroV)
mit Cafeteria roenbergensis virus BV-PW1
- Spezies Rheavirus sinusmexicani (Cafeteria-roenbergensis-Virus, CroV)
- Klade (Gattung?) „Namaovirus“[12] („Sturgeon Nucleocytoplasmic Large DNA Virus“, sNCLDV)[13][14][15][16]
- Spezies „Namao-Virus“ (en. „Namao virus“, NV)
- ohne Gattungszuweisung
- Spezies „Faunusvirus sp.“ (zu unterscheiden von der offiziell bestätigten Gattung Faunusvirus in der Familie Chaseviridae, Klasse Caudoviricetes)[17][18][5][19]
- Spezies „cPacV 1605“[20][21]
- Spezies „mPacV 611“[20][22]
- Gattung Rheavirus (früher Cafeteriavirus)
- Unterfamilie Klosneuvirinae (Klosneuviren, engl. Klosneuviruses)[23]
- Gattung Fadolivirus
- Gattung Theiavirus (mit Bodo-saltans-Virus, BsV)
- Gattung Yasminevirus
- Gattung „Catovirus“ (CatV)
- Gattung „Klosneuvirus“ (KlosnV)
- …
- Unterfamilie Aliimimivirinae (Mimiviridae Gruppe II, Cafeteriaviren)[11]
- Unterfamilie Megamimivirinae[18] (Mimiviridae Gruppe I, Mimiviren s. l., früher auch „Mimivirinae“ genannt[5])
- Gattung Cotonvirus[24]
- Gattung Mimivirus[25] (Mimiviridae: Gruppe I Linie A, MA[26])
- Gattung Megavirus (Mimiviridae: Gruppe I Linie C, MC,[26] Courdo11-Linie oder Megavirus-Linie[27])
- Gattung Moumouvirus (Mimiviridae: Gruppe I Linie B, MB,[26] Moumouvirus-Linie)
- Gattung Tupanvirus (Mimiviridae Gruppe I, Tupanvirus-Linie)
- Spezies Tupanvirus altamarinense mit
Tupanvirus deep ocean - Spezies Tupanvirus salinum mit
Tupanvirus soda lake
- Spezies Tupanvirus altamarinense mit
- Gattung „Platanovirus“ (Mimiviridae Gruppe I, Tupanvirus-Linie)[28][29][30]
- Gattung „Satyrvirus“ (Mimiviridae Gruppe I, Tupanvirus-Linie)[18][5][19][31]
- ohne Gattungszuweisung:
- ohne nähere Zuweisung (basal im Mimiviridae)
- Klade – ursprünglich als Unterfamilie „Aquavirinae“ („Aquaviren“) vorgeschlagen[6][7] (nicht realisiert)
Anm.:
- Bei den Spezies (bzw. Stämmen) der drei Gattungen Mimivirus, Moumouvirus und Megavirus kann zur Unterscheidung dem abgekürzten Gattungsnamen der tiefergestellte Buchstabe der jeweiligen Linie (A, B bzw. C) beigefügt werden: MA. für Mimivirus, MB für Moumouvirus und MC für Megavirus.[26]
- T – bei Namensgleichheit mit der Spezies deutet ein hochgestelltes ‚T‘ einen Typus- oder Referenzstamm an.
Die Phylogenie der Mimiviridae ist nach Aylward et al. (2021) vereinfacht wie folgt:[4]
| Mimiviridae |
| ||||||||||||||||||||||||||||||||||||||||||
Die genauen Verwandtschaftsverhältnisse sind aber möglicherweise noch in der Diskussion, nach Schulz et al. (2018) steht z. B. die Linie A (Gattung Mimivirus) basal in der Gruppe I, nicht die Tupanviren (Gattung Tupanvirus).[18] Dies könnte ein Grund sein, Mimivirus zunächst noch keiner Unterfamilie zuzuordnen.
Megamimivirinae (inkl. Mimivirus): Mimiviridae Gruppe I
Diese Gruppe umfasst die Mimiviren im weiteren Sinn. Das erste entdeckte Mitglied der Familie Mimiviridae, die Gattung Mimivirus mit der Art Acanthamoeba polyphaga Mimivirus (ApMV, auch APMV, offiziell Mimivirus bradfordmassiliense) wurde bereits 2003 entdeckt.[36]
Mit der Einrichtung einer Unterfamilie Megamimivirinae folgte das ICTV Vorschlägen etwa von Schulz et al. (2018),[18] gelegentlich wurde auch als kürzerer Name „Mimivirinae“[5] oder „Megavirinae“[6][7] vorgeschlagen.
Weitere Mitglieder der Unterfamilie, die ursprünglich auch in der Gattung Mimivirus subsumiert waren,[7][37] sind:
- Gattung Mimivirus (früher: Linie A, Mimiviren im engeren Sinn) – übriggeblieben nach Abtrennung der Linien B und C in eigene Gattungen, enthält den ursprünglichen Vertreter ApMV sowie AcMV.
- Gattung Moumouvirus (früher: Linie B)
- Gattung Megavirus (früher: Linie C oder Courdo11-Gruppe)
Zu der in der Unterfamilie Megamimivirinae zusammengefassten Verwandtschaft der Gattung Mimivirus gehören auch die später entdeckten Gattungen Tupanvirus,[18][30][38][39][40] Cotonvirus, sowie (per Vorschlag) „Satyrvirus“ und „Platanovirus“.[29][30]
Aliimimivirinae: Mimiviridae Gruppe II (Cafeteriaviren)
Diese Gruppe beinhaltet die vom ICTV bereits mit Einrichtung der Familie Mimiviridae bestätigte Spezies Cafeteria-roenbergensis-Virus (offiziell: Rheavirus sinusmexicani mit Gattung Rheavirus – ursprünglich Cafeteriavirus genannt). Dies ist der bislang (Stand Ende April 2023) einzige offiziell bestätigte Vertreter dieser Unterfamilie. Es gibt jedoch eine Reihe weitere Vorschläge für die Klade Mimiviridae Gruppe II.[11]bzw. Unterfamilie Aliimimivirinae.
Im Februar 2013 schlugen S. Clouthier et al. für das von ihnen gefunden „Namao-Virus“ (NV), das Störe (englisch sturgeons) wie den See-Stör (Acipenser fulvescens) parasitiert, eine Gruppe vor mit der provisorischen Bezeichnung „sturgeon Nucleocytoplasmic Large DNA Virus“ (sNCLDV). NV konnte innerhalb der Mimiviridae zunächst keiner bekannten Gruppe zugeordnet werden. Phylogenetische Analysen zeigten aber, dass NV und das Cafeteria-roenbergensis-Virus (CroV) Schwestertaxa sein sollten.[13][14][15][16] Die Klade mit NV wird von Schulz et al. (2020) informell als Namaoviruses ‚Namaoviren‘ bezeichnet.[12]
Klosneuvirinae: Klosneuviren
Eine weitere Klade der Mimiviridae sind die 2017 zunächst nur durch Metagenomik entdeckten Klosneuviren mit den Kandidatengattungen „Klosneuvirus“, „Catovirus“, „Hokovirus“[23][4] und „Indivirus“.[23] Das ICTV folgte einem Vorschlag, wie er von Schulz et al. (2017) und Rolland et al. (2018) vorgetragen wurde, diese in einer Unterfamilie Klosneuvirinae zusammenzufassen.[23][27][1]
Die Gruppe umfasst auch später hinzugekommene Vertreter, insbesondere Bodo-saltans-Virus (BsV, offiziell Theiavirus salishense). Das ICTV hat diese Art in eine eigene Gattung Theiavirus gestellt, abgetrennt von der Gattung Klosneuvirus (anders als in der Taxonomie NCBI).[41] Weitere Mitglieder der Klosneuviren-Unterfamilie sind die beiden Gattungen Fadolivirus[42] und Yasminevirus[43] Stämme dieser drei Arten aus konnten unter Zuhilfenahme geeigneter Wirtszellen kultiviert werden.[27]
Ebenfalls in die Verwandtschaft der Klosneuviren passen zwei Contigs (LCMiAC01[44] und LCMiAC02[45]) aus Metagenomanalysen des Schwarzen Rauchers „Lokis Schloss“ (englisch Loki’s Castle), Namensgeber der Archaeengruppe Lokiarchaeota.[46]
„gvSAG AB-566-O17“
Der von Wilson et al. (2017) beschriebene Kandidat „gvSAG AB-566-O17“ (vom NCBI als nicht näher klassifizierte Mimiviridae-Spezies mit Mimivirus AB-566-O17 bezeichnet[34]) ist als Kandidat der Mimiviridae ein „Mimivirus-like virus“. Nach den Autoren (Fig. 2) ist dieses Virus mit ApMV (d. h. der Gattung Mimivirus) weitläufiger verwandt als CroV (der Gattung Rheavirus, früher Cafeteriavirus), aber näher als PgV-16T (Spezies Tethysvirus hollandense, Mesomimiviridae) oder AaV (Spezies Kratosvirus quantuckense, Schizomimiviridae); es ist daher keiner dieser anderen Imitervirales-Familien zuzuordnen. Dieses Virus steht daher entweder basal in den Mimivirdae (wenn man diese Familie weit genug fasst) oder bildet ein Schwestertaxon zu diesen.[33]
Mimiviridae aus Waldbodenproben
Im November 2018 berichteten Frederik Schulz et al. über die Entdeckung von 16 neuen Riesenviren per Metagenomanalyse aus Waldbodenproben. Für diese Viren wurden vorläufige Namen vergeben, die meist auf ihre Herkunft hinweisen. Zur Familie der Mimiviridae gehören darunter vorschlagsgemäß die folgenden:[18][5][19]
- Megamimivirinae inkl. Mimivirus (Mimiviren im weiteren Sinn, Gruppe I):
„Satyrvirus sp.“ (Tupanvirus),[31] - Aliimimivirinae (Cafeteriaviren, Gruppe II):
„Faunusvirus sp.“ (nicht zu verwechseln mit der offiziellen Gattung Faunusvirus der Chaseviridae),[17] - Klosneuvirinae (Klosneuviren):
„Gaeavirus sp.“,[47] „Homavirus sp.“,[48] „Barrevirus sp.“,[49] „Dasosvirus sp.“,[50] „Edafosvirus sp.“,[51] „Terrestrivirus sp.“,[52] „Harvfovirus sp.“[53] und „Hyperionvirus sp.“.[54]
Merkmale der Mimiviridae im Vergleich
| Virus | Aminoacyl-tRNA-Synthetase | Octocorallia-| Proteinfilamente (Länge) | Stargate | Bekannter Virophage | Cytoplasmische | Virion-Fabrik Wirt | Megavirus chilensis (MVc) | 7 (Tyr, Arg, Met, Cys, Trp, Asn, Ile) | ja | ja (75 nm) | ja | nein | ja | Acanthamöben (Unikonta, Amoebozoa) | Mamavirus AcMV | 4 (Tyr, Arg, Met, Cys) | ja | ja (120 nm) | ja | ja | ja | Acanthamöben (Unikonta, Amoebozoa) | Mimivirus ApMV | (Wildtyp M1) 4 (Tyr, Arg, Met, Cys) | ja | ja (120 nm) | ja | ja | ja | Acanthamöben (Unikonta, Amoebozoa) | Mimivirus M4 | (bald/fiberless Variante) 2 (Met, Cys) | nein | nein | ja | resistent | ja | Acanthamöben (Unikonta, Amoebozoa) | Cafeteria-roenbergensis-Virus (CroV) | 1 (Ile) | ja | nein | nein | ja | ja | Phagotrophische Protozoen (Heterokonta, Stramenopiles) | |
|---|
| Virus | Genom-Größe (bp) | Gene | Kapsid-Durchmesser (nm) | Genbank-Nr. |
|---|---|---|---|---|
| Megavirus chilensis (MVC)[57] | 1.259.197 | 1120 Proteine (abgeleitet) | 440 | JN258408 |
| Mamavirus (AMV)[58] | 1.191.693 | 1023 Proteine (abgeleitet) | 390 | JF801956 |
| Mimivirus ApMV[59][60] (Wildtyp M1) | 1.181.549 | 979 Proteine (39 nicht-codierend) | 390 | NC_014649 |
| Mimivirus M4[61] (bald/fiberless Variante) | 981.813 | 756 Proteine (abgeleitet) | 390 | JN036606 |
| Cafeteria-roenbergensis-Virus (CroV)[62] | 617.453 (730 kbp) | 544 Proteine (abgeleitet) | 300 | NC_014637 |
Virophagen
Es gibt Satellitenviren, die den Syntheseapparat der Mimiviridae (und vermutlich auch anderer Viren der Klasse Megaviricetes[27]) für ihre eigene Vermehrung nutzen und Virophagen genannt werden, wenn sie ihren Helferviren (Wirtsviren) schaden. Virophagen replizieren natürlich nicht im Virion des Wirtsvirus, weil dieses keinen Stoffwechsel hat. Stattdessen nutzen sie den durch das Wirtsvirus umgestalteten Proteinsyntheseapparat der Wirtszelle (das Viroplasma) und sind von den Replikationsenzymen des Wirtsvirus abhängig. Die Virophagen der Riesenviren sind im Vergleich zu Satellitenviren anderer Helferviren ebenfalls vergleichsweise riesig und haben auch ein komplexeres Genom.[63][64]
- Der zuerst entdeckte Fall dieser Art ist das Sputnikvirus (offiziell Mimivirus-dependent virus Sputnik), als dessen Wirtsvirus das Mimivirus ApMV dienen kann.
- Ein weiterer Virophage namens Zamilon (offiziell Mimivirus-dependent virus Zamilon) befällt nur die Megamimivirinae-Linien B und C (d. h. die Gattungen Moumouvirus respektive Megavirus), während der Virophage Sputnik 3 diese und auch Linie A (Gattung Mimivirus) befällt, darunter das Mamavirus getauften Mimivirus.[37] Inzwischen ist bekannt, dass die Linien B und C von Zamilon befallen werden, die Linie A jedoch eine MIMIVIRE (englisch mimivirus virophage resistance element) genannte Resistenz gegen Zamilon, nicht aber gegen Sputnik 3, aufweisen. MIMIVIRE funktioniert ähnlich wie das CRISPR/Cas-System.[65][66]
Etymologie
Der Name der Familie Mimiviridae leitet sich ab vom Gattungsnamen Mimivirus des zuerst gefundenen Vertreters (ApMV, Acanthamoeba polyphaga mimivirus), dieser ist eine Zusammenziehung aus englisch mimicking microbe ‚eine Mikrobe nachahmend‘, da man diese Viren zunächst für Bakterien (Kokken) gehalten hatte. Angehängt ist der Suffix für Virenfamilien, ‚-viridae‘.
Der Name der Unterfamilie Megamimivirinae leitet sich ab von der Familie Mimiviridae, zu der sie gehört, und der enthaltenen Gattung Megavirus, was auch ein Hinweis auf die Größe dieser Viren ist. Angehängt ist wie bei den anderen Unterfamilien der Suffix für Virenunterfamilien, ‚-virinae‘.
Der Name der Unterfamilie Klosneuvirinae leitet sich ab von Klosterneuburg bei Wein, wo der erste Vertreter, die (mit Stand 1. Mai 2023) noch nicht vom ICTV bestätigte Spezies „Klosneuvirus KNV1“ gefunden wurde.
Der Namen der Unterfamilie Aliimimivirinae leitet sich ab von lateinisch alii- ‚anders-‘, was darauf hinweist dass diese Viren einen Zweig der Mimiviridae bilden, der zu den näheren Verwandten der namensgebenden Gattung Mimivirus noch hinzukommt,[4] was der basalen Position im phylogenetischen Baum der Mimiviridae geschuldet sein mag.
Weblinks
- Ana Cláudia dos S. P. Andrade, Thalita S. Arantes, Rodrigo A. L. Rodrigues, Talita B. Machado, Fábio P. Dornas, Melissa F. Landell, Cinthia Furst, Luiz G. A. Borges, Lara A. L. Dutra, Gabriel Almeida, Giliane de S. Trindade, Ivan Bergier, Walter Abrahão, Iara A. Borges, Juliana R. Cortines, Danilo B. de Oliveira, Erna G. Kroon, Jônatas S. Abrahão: Ubiquitous giants: a plethora of giant viruses found in Brazil and Antarctica, in: Virology Journal, Band 15, Nr. 22, 24. Januar 2018, doi:10.1186/s12985-018-0930-x
- Christelle Desnues, Bernard La Scola, Natalya Yutin, Ghislain Fournous, Catherine Robert, Saïd Azza, Priscilla Jardot, Sonia Monteil, Angélique Campocasso, Eugene V. Koonin, and Didier Raoult: Provirophages and transpovirons as the diverse mobilome of giant viruses, in: PNAS 109 (44) vom 30. Oktober 2012, S. 18078–18083, doi:10.1073/pnas.1208835109
Anmerkungen
- ↑ Die Supergruppe der Nucleocytoplasmic large DNA viruses (NCLDVs) war zunächst – noch vor der Einrichtung höherer taxonomischer Rangstufen (als der Ordnung) durch das ICTV – als Ordnung „Megavirales“ (s. l.) vorgeschlagen worden.
- ↑ Deeg et al. (2018) hatten umgekehrt die Familie Mimiviridae mit ApMV und CroV herabgestuft zu einer Unterfamilie „Megavirinae“, in der die Klosneuviren und die Mimiviridae Gruppe I Schwesterkladen ohne Rang darstellten. In der umfassenden Familie Mimiviridae wäre dann noch Platz gewesen für weitere Mitglieder und Unterfamilien.[5] Das ICTV ist dieser Idee nicht gefolgt, sondern hat die weitere Verwandtschaft in Schwesterfamilien der Mimiviridae innerhalb der Imitervirales untergebracht.[1]
Andere Autoren hatten aufgrund der etwas engeren Verwandtschaft der Klosneuviren mit den Cafeteriaviren[4] (inkl. „Namao-Virus“) eine gemeinsame Unterfamilie „Aquavirinae“ innerhalb der Mimiviridae vorgeschlegen.[6][7] Das ICTV hat jedoch für beide Gruppen eigene Unterfamilien eingerichtet.
Ein weiterer früherer Vorschlag war eine umfassende Familie „Megaviridae“, diese Funktion übernimmt jetzt schon die die Ordnung Imitervirales.[8] . - ↑ englisch Molecular phylogenetic analysis of family Mimiviridae based on the concatenated NCLDV core genes. Terminologie angepasst an ICTV Master Species List #38, 8. April 2023.
Einzelnachweise
- ↑ a b c d e f ICTV: Master Species Lists § ICTV Master Species List 2022 MSL38 v1 (xlsx), 8. April 2023.
- ↑ a b c d e f ICTV: ICTV Master Species List 2019.v1, New MSL including all taxa updates since the 2018b release, March 2020 (MSL #35)
- ↑ Eugene V. Koonin, Natalya Yutin: Evolution of the Large Nucleocytoplasmatic DNA Viruses of Eukaryotes and Convergent Origins of Viral Gigantism. In: Advances in Virus research, Band 103, AP 21. Januar 2019, doi:10.1016/bs.aivir.2018.09.002, S. 167–202. Die Klosneuviren sind teilweise als Klosneviruses fehlgeschrieben.
- ↑ a b c d e f g h Frank O. Aylward, Jônatas S. Abrahão, Corina P. D. Brussaard C, Matthias G. Fischer, Mohammad Moniruzzaman, Hiroyuki Ogata, Curtis A. Suttle: Create 3 new families, 3 subfamilies, 13 genera, and 20 new species within the order Imitervirales (phylum Nucleocytoviricota) and rename two existing species (zip:docx). Vorschlag 2022.004F an das ICTV vom Oktober 2021.
Anm.: Entgegen Vorschlag (Tbl. 1) wurde die Gattung Mimivirus nicht in die Unterfamilie Megamimivirinae aufgenommen. Phylogenetische Analysen zeigten zu deren Mitgliedern einen größeren Abstand als diese untereinander. - ↑ a b c d e f Christoph M. Deeg, Cheryl-Emiliane T. Chow, Curtis A. Suttle: The kinetoplastid-infecting Bodo saltans virus (BsV), a window into the most abundant giant viruses in the sea… In: eLife Sciences, 7, März 2018; doi:10.7554/eLife.33014, ResearchGate.
- ↑ a b c d List of the main “giant” viruses known as of today. (PDF) Centre national de la recherche scientifique, Université d’Aix-Marseille, 18. April 2018.
- ↑ a b c d e List of the main “giant” viruses known as of today. (PDF) Centre national de la recherche scientifique, Université d’Aix-Marseille, März 2019.
- ↑ Natalya Yutin, Philippe Colson, Didier Raoult, Eugene V. Koonin: Mimiviridae: clusters of orthologous genes, reconstruction of gene repertoire evolution and proposed expansion of the giant virus family. In: BMC: Virology Journal, Band 10, Nr. 107, 4. April 2013; doi:10.1186/1743-422X-10-106, PMC 3620924 (freier Volltext), PMID 23557328.
- ↑ Haruna Takahashi, Sho Fukaya, Chihong Song, Kazuyoshi Murata, Masaharu Takemura: Morphological and Taxonomic Properties of the Newly Isolated Cotonvirus japonicus, a New Lineage of the Subfamily Megavirinae. In: ASM Journals: Journal of Virology, Band 95, Nr. 18, 25. August 2021; doi:10.1128/JVI.00919, ResearchGate.
- ↑ NCBI Taxonomy Browser: Search for …, Nucleotide: Search.
- ↑ a b V. M. Marcelino, M. V. P. C. Espinola, V. Serrano-Solis, S. T. Farias: Evolution of the genus Mimivirus based on translation protein homology and its implication in the tree of life. In: Genet.Mol.Res., 27. September 2017, Band 16, Nr. 3, S. gmr16039784, doi:10.4238/gmr16039784.
- ↑ a b c Frederik Schulz, Simon Roux, David Paez-Espino, Sean Jungbluth, David A. Walsh, Vincent J. Denef, Katherine D. McMahon, Konstantinos T. Konstantinidis, Emiley A. Eloe-Fadrosh, Nikos C. Kyrpides, Tanja Woyke: Giant virus diversity and host interactions through global metagenomics. In: Nature, Band 578, S. 432–436, 22. Januar 2020, doi:10.1038/s41586-020-1957-x, PMID 31968354, PMC 7162819 (freier Volltext), insbes. Fig. 1 und Supplementary Data 1: Maximum likelihood phylogeny and genome features of superclades SC1-SC10 (Fig. SC6 zu GVMAG-S-1014582-52).
- ↑ a b Sharon C. Clouthier, E. Vanwalleghem, S. Copeland, C. Klassen, G. Hobbs, O. Nielsen, Eric D. Anderson: A new species of nucleo-cytoplasmic large DNA virus (NCLDV) associated with mortalities in Manitoba lake sturgeon Acipenser fulvescens. In: Dis Aquat Organ., 102(3), 28. Februar 2013, S. 195–209; doi:10.3354/dao02548, PMID 23446969.
- ↑ a b Sharon C. Clouthier, E. Vanwalleghem, Eric D. Anderson: Sturgeon nucleo-cytoplasmic large DNA virus phylogeny and PCR tests. In: Dis Aquat Organ. Band 117, Nr. 2, 9. Dezember 2015, S. 93–106; doi:10.3354/dao02937, PMID 26648102.
- ↑ a b Sharon C. Clouthier, Eric D. Anderson, Gael Kurath, Rachel B. Breyta: Molecular systematics of sturgeon nucleocytoplasmic large DNA viruses. In: Mol Phylogenet Evol 128, Juli 2018; doi:10.1016/j.ympev.2018.07.019, ResearchGate.
- ↑ a b Jean-Michel Claverie, Chantal Abergel: Mimiviridae: An Expanding Family of Highly Diverse Large dsDNA Viruses Infecting a Wide Phylogenetic Range of Aquatic Eukaryotes. In: Viruses, Band 10, Nr. 9, 10(9), 18. September 2018, S. 506; doi:10.3390/v10090506, PMC 6163669 (freier Volltext), PMID 30231528.
- ↑ a b NCBI Taxonomy Browser: Faunusvirus sp. (species).
- ↑ a b c d e f g h Frederik Schulz, Lauren Alteio, Danielle Goudeau, Elizabeth M. Ryan, Feiqiao B. Yu, Rex R. Malmstrom, Jeffrey Blanchard, Tanja Woyke: Hidden diversity of soil giant viruses. In: Nature Communications, Band 9, Nr. 4881, 19. November 2018, doi:10.1038/s41467-018-07335-2, PMID 30451857, PMC 6243002 (freier Volltext).
- ↑ a b c Vincent Racaniello, David Tuller, Gertrud U. Rey: Bodo saltans virus, an abundant giant aquatic Mimivirus. Virology blog, 28. Dezember 2017
- ↑ a b David M. Needham, Susumu Yoshizawa, Toshiaki Hosaka, Camille Poirier, Chang Jae Choi, Elisabeth Hehenberger, Nicholas A. T. Irwin, Susanne Wilken, Cheuk-Man Yung, Charles Bachy, Rika Kurihara, Yu Nakajima, Keiichi Kojima, Tomomi Kimura-Someya, Guy Leonard, Rex R. Malmstrom, Daniel R. Mende, Daniel K. Olson, Yuki Sudo, Sebastian Sudek, Thomas A. Richards, Edward F. DeLong, Patrick J. Keeling, Alyson E. Santoro, Mikako Shirouzu, Wataru Iwasaki, Alexandra Z. Worden: A distinct lineage of giant viruses brings a rhodopsin photosystem to unicellular marine predators. In: PNAS, 23. September 2019, ISSN 0027-8424; doi:10.1073/pnas.1907517116, inklusive Supplement 1 (xlsx).
- ↑ NCBI Assembly: cPacV-1605, BioSample: DN-24_S10. Koordinaten: 36,688° N, 122,386° W (Monterey Bay).
- ↑ NCBI Nucleotide: Search: mPacV-611, BioSample: Marine eukaryotic communities from CALCOFI LINE 67, Pacific Ocean - C012_Sort48C_1. Koordinaten: 36,1443° N, 122,57° W (Monterey Bay).
- ↑ a b c d Frederik Schulz, Natalya Yutin, Natalia N. Ivanova, Davi R. Ortega, Tae Kwon Lee, Julia Vierheilig, Holger Daims, Matthias Horn, Michael Wagner: Giant viruses with an expanded complement of translation system components. In: Science. Band 356, Nr. 6333, 7. April 2017, ISSN 0036-8075, S. 82–85, doi:10.1126/science.aal4657, PMID 28386012, bibcode:2017Sci...356...82S (englisch, Online)., UCPMS ID: 1889607, escholarship.org (PDF; 1,8 MB).
- ↑ Bruna Luiza de Azevedo, João Pessoa Araújo Júnior, Leila Sabrina Ullmann, Rodrigo Araújo Lima Rodrigues, Jônatas Santos Abrahão: The Discovery of a New Mimivirus Isolate in Association with Virophage-Transpoviron Elements in Brazil Highlights the Main Genomic and Evolutionary Features of This Tripartite System. In: MDPI: Viruses, Band 14, Nr. 2, Section General Virology, 21. Januar 2022, S. 206; doi:10.3390/v14020206.
- ↑ ICTV Taxon Details: Genus: Mimivirus.
- ↑ a b c d Sandra Jeudy, Lionel Bertaux, Jean-Marie Alempic, Audrey Lartigue, Matthieu Legendre, Lucid Belmudes, Sébastien Santini, Nadège Philippe, Laure Beucher, Emanuele G. Biondi, Sissel Juul, Daniel J. Turner, Yohann Couté, Jean-Michel Claverie, Chantal Abergel: Exploration of the propagation of transpovirons within Mimiviridae reveals a unique example of commensalism in the viral world. In: The ISME Journal, Band 14, S. 727–739, 10. Dezember 2019; doi:10.1038/s41396-019-0565-y, PMID 31822788, PMC 7031253 (freier Volltext).
- ↑ a b c d Clara Rolland, Julien Andreani, Amina Cherif Louazani, Sarah Aherfi, Rania Francis, Rodrigo Rodrigues, Ludmila Santos Silva, Dehia Sahmi, Said Mougari, Nisrine Chelkha, Meriem Bekliz, Lorena Silva, Felipe Assis, Fábio Dornas, Jacques Yaacoub Bou Khalil, Isabelle Pagnier, Christelle Desnues, Anthony Levasseur, Philippe Colson, Jônatas Abrahão, Bernard La Scola: Discovery and Further Studies on Giant Viruses at the IHU Mediterranee Infection That Modified the Perception of the Virosphere. In: Viruses, 11(4), März/April 2019, pii: E312, doi:10.3390/v11040312, PMC 6520786 (freier Volltext), PMID 30935049. Anm.: Die „Klosneuvirinae“ sind per Namensendung eine (vorgeschlagene) Unterfamilie der Mimiviridae, erst recht können die vorgeschlagenen Vertreter Yasminevirus und Fadolivirus nicht auch selbst neue Familien repräsentieren. Sie sind nicht die ersten isolierten Vertreter dieser Unterfamilie, das ist – wie an anderer stelle korrekt festgestellt wird, Bodo-saltans-Virus.
- ↑ Rolf Michel, Liane Junglas, Silke Loch, Claudia Wylezich, Karl-Dieter Müller, Bärbel Hauröder: Experimental co-infection of Saccamoeba lacustris with Mimivirus-like Giant virus and a small Satellite virus, in: Endocytobiosis and Cell Research, Band 29, 15. Mai 2018, S. 1–6, ResearchGate, thulb.
- ↑ a b David M. Needham, Susumu Yoshizawa, Toshiaki Hosaka, Camille Poirier, Chang Jae Choi, Elisabeth Hehenberger, Nicholas A. T. Irwin, Susanne Wilken, Cheuk-Man Yung, Charles Bachy, Rika Kurihara, Yu Nakajima, Keiichi Kojima, Tomomi Kimura-Someya, Guy Leonard, Rex R. Malmstrom, Daniel R. Mende, Daniel K. Olson, Yuki Sudo, Sebastian Sudek, Thomas A. Richards, Edward F. DeLong, Patrick J. Keeling, Alyson E. Santoro, Mikako Shirouzu, Wataru Iwasaki, Alexandra Z. Worden: A distinct lineage of giant viruses brings a rhodopsin photosystem to unicellular marine predators. In: PNAS, 23. September 2019, doi:10.1073/pnas.1907517116, ISSN 0027-8424.
- ↑ a b c Jônatas Abrahão, Lorena Silva, Ludmila Santos Silva, Jacques Yaacoub Bou Khalil, Rodrigo Rodrigues, Thalita Arantes, Felipe Assis, Paulo Boratto, Miguel Andrade, Erna Geessien Kroon, Bergmann Ribeiro, Ivan Bergier, Herve Seligmann, Eric Ghigo, Philippe Colson, Anthony Levasseur, Guido Kroemer, Didier Raoult, Bernard La Scola: Tailed giant Tupanvirus possesses the most complete translational apparatus of the known virosphere. In: Nature Communications. Band 9, Nr. 1, 27. Februar 2018, doi:10.1038/s41467-018-03168-1 (Online).
- ↑ a b NCBI Taxonomy Browser: Satyrvirus sp. (species).
- ↑ NCBI Nucleotide: MAG: viral metagenome isolate GVMAG-S-1014582-52, …
- ↑ a b William H. Wilson, Ilana C. Gilg, Mohammad Moniruzzaman, Erin K. Field, Sergey Koren, Gary R. LeCleir, Joaquín Martínez Martínez, Nicole J. Poulton, Brandon K. Swan, Ramunas Stepanauskas, Steven W. Wilhelm: Genomic exploration of individual giant ocean viruses. In: ISME Journal, 11(8), August 2017, S. 1736–1745, doi:10.1038/ismej.2017.61, PMC 5520044 (freier Volltext), PMID 28498373, nature, PDF (746 kB) Associated Data (Supplements): Supplementary Dataset 1 (gvSAG AB-572-A11), Fig. 2 (gvSAG AB-566-O17).
- ↑ a b NCBI Taxonomy Browser: Mimivirus AB-566-O17 (Acronym: gvSAG AB-566-O17, gvSAG_O17) (species).
- ↑ NCBI: Faunusvirus sp. (species).
- ↑ Marie Suzan-Monti, Bernard La Scola, Didier Raoult: Genomic and evolutionary aspects of Mimivirus. In: Virus Research, Band 117, Nr. 1, April 2006, S. 145–155, doi:10.1016/j.virusres.2005.07.011 , PMID 16181700.
- ↑ a b Morgan Gaia, Samia Benamar, Mondher Boughalmi, Isabelle Pagnier, Olivier Croce, Philippe Colson, Didier Raoult, Bernard La Scola: Zamilon, a Novel Virophage with Mimiviridae Host Specificity. In: PLoS One. Band 9(4), 2014, S. e94923, doi:10.1371/journal.pone.0094923, PMID 24747414, PMC 3991649 (freier Volltext). Epub 18. April 2014
- ↑ Daniel Lingenhöhl: Verschieben neue Riesenviren die Grenzen des Lebens? Spektrum.de, 28. Februar 2018
- ↑ Nadja Podbregar: Ungewöhnliche Riesenviren entdeckt. wissenschaft.de, 27. Februar 2018
- ↑ Riesenviren an der Grenze zum Leben, auf: scinexx.de, 28. Februar 2018
- ↑ NCBI Taxonomy Browser: Bodo saltans virus (spezies).
- ↑ Hadjer Boudjemaa, Julien Andreani, Idir Bitam, Bernard La Scola: Diversity of Amoeba-Associated Giant Viruses Isolated in Algeria. In: Diversity, Band 12, Nr. 6, 215, 29. Mai 2020, doi:10.3390/d12060215.
- ↑ Leena Hussein Bajrai, Saïd Mougari, Julien Andreani, Emeline Baptiste, Jeremy Delerce, Didier Raoult, Esam Ibraheem Azhar, Bernard La Scola, Anthony Levasseur: Isolation of Yasminevirus, the First Member of Klosneuvirinae Isolated in Coculture with Vermamoeba vermiformis, Demonstrates an Extended Arsenal of Translational Apparatus Components. In: JVirol, Band 94, Nr. 1, Herbst 2019; doi:10.1128/JVI.01534-19, ResearchGate.
- ↑ NCBI Taxonomy Browser: Mimivirus LCMiAC01 (species). Nucleotide: MAG: Mimivirus LCMiAC01….
- ↑ NCBI Taxonomy Browser: Mimivirus LCMiAC02 (species). Nucleotide: MAG: Mimivirus LCMiAC02….
- ↑ Disa Bäckström, Natalya Yutin, Steffen L. Jørgensen, Jennah Dharamshi, Felix Homa, Katarzyna Zaremba-Niedwiedzka, Anja Spang, Yuri I. Wolf, Eugene V. Koonin, Thijs J. G. Ettema: Virus Genomes from Deep Sea Sediments Expand the Ocean Megavirome and Support Independent Origins of Viral Gigantism. In: mBio, Band 10, Nr. 2, 2019; doi:10.1128/mBio.02497-18.
- ↑ NCBI Taxonomy Browser: Gaeavirus sp. (species).
- ↑ NCBI Taxonomy Browser: Homavirus sp. (species).
- ↑ NCBI Taxonomy Browser: Barrevirus sp. (species).
- ↑ NCBI Taxonomy Browser: Dasosvirus (species).
- ↑ NCBI Taxonomy Browser: Edafosvirus sp. (species).
- ↑ NCBI Taxonomy Browser: Terrestrivirus sp. (species).
- ↑ NCBI Taxonomy Browser: Harvfovirus (species).
- ↑ NCBI Taxonomy Browser: Hyperionvirus sp. (species).
- ↑ Autor=Nathan Zauberman, Y. Mutsafi, D. B. Halevy, E. Shimoni, E. Klein, C. Xiao, S. Sun, A. Minsky: Distinct DNA exit and packaging portals in the virus Acanthamoeba polyphaga mimivirus. In: PLoS Biol. Band 6, Nr. 5, 2008, S. e114, doi:10.1371/journal.pbio.0060114, PMID 18479185, PMC 2430901 (freier Volltext).
- ↑ M. G. Fischer, C. A. Suttle: A Virophage at the Origin of Large DNA Transposons. In: Science. Band 332, Nr. 6026, 2011, S. 231–234, doi:10.1126/science.1199412, PMID 21385722.
- ↑ Defne Arslan, Matthieu Legendre, Virginie Seltzer, Chantal Abergel, Jean-Michel Claverie: Distant Mimivirus relative with a larger genome highlights the fundamental features of Megaviridae. In: PNAS. 10. Oktober 2011, doi:10.1073/pnas.1110889108.
- ↑ P. Colson u. a.: Viruses with More Than 1,000 Genes: Mamavirus, a New Acanthamoeba polyphaga mimivirus Strain, and Reannotation of Mimivirus Genes. In: Genome Biology and Evolution. Band 3, 2011, S. 737–742, doi:10.1093/gbe/evr048, PMID 21705471, PMC 3163472 (freier Volltext).
- ↑ Didier Raoult, Stéphane Audic, Catherine Robert, Chantal Abergel, Patricia Renesto, Hiroyuki Ogata, Bernard La Scola, Marie Suzan, Jean-Michel Claverie: The 1.2-Megabase Genome Sequence of Mimivirus. In: Science. Band 306, Nr. 5700, 19. November 2004, S. 1344–1350, doi:10.1126/science.1101485, PMID 15486256.
- ↑ Matthieu Legendre u. a.: Breaking the 1000-gene barrier for Mimivirus using ultra-deep genome and transcriptome sequencing. In: Virology Journal. Band 8, 2011, S. 99, doi:10.1186/1743-422X-8-99, PMID 21375749, PMC 3058096 (freier Volltext).
- ↑ Mickaël Boyer, Saïd Azza, Lina Barrassi, Thomas Klose, Angélique Campocasso, Isabelle Pagnier, Ghislain Fournous, Audrey Borg, Catherine Robert, Xinzheng Zhang, Christelle Desnues, Bernard Henrissat, Michael G. Rossmann, Bernard La Scola, Didier Raoult: Mimivirus shows dramatic genome reduction after intraamoebal culture. In: Proceedings of the National Academy of Sciences. Band 108, Nr. 25, 2011, S. 10296–10301, doi:10.1073/pnas.1101118108, PMC 3121840 (freier Volltext).
- ↑ M. G. Fischer u. a.: Giant virus with a remarkable complement of genes infects marine zooplankton. In: Proceedings of the National Academy of Sciences. Band 107, Nr. 45, 2010, S. 19508–19513, doi:10.1073/pnas.1007615107, PMC 2984142 (freier Volltext).
- ↑ David Paez-Espino, Jinglie Zhou, Simon Roux, Stephen Nayfach, Georgios A. Pavlopoulos, Frederik Schulz, Katherine D. McMahon, David Walsh, Tanja Woyke, Natalia N. Ivanova, Emiley A. Eloe-Fadrosh, Susannah G. Tringe, Nikos C. Kyrpides: Diversity, evolution, and classification of virophages uncovered through global metagenomics. In: Microbiome, Band 7, Nr. 157, 10. Dezember 2019, doi:10.1186/s40168-019-0768-5.
- ↑ Meriem Bekliz, Philippe Colson, Bernard La Scola: The Expanding Family of Virophages. In: MDPI: Viruses, Band 8, Nr. 11, Special Issue Viruses of Protozoa, 23. November 2016, 317; doi:10.3390/v8110317.
- ↑ Anthony Levasseur, Meriem Bekliz, Eric Chabrière, Pierre Pontarotti, Bernard La Scola, Didier Raoult: MIMIVIRE is a defence system in mimivirus that confers resistance to virophage. In: Nature, 2016, doi:10.1038/nature17146, PMID 26934229, ResearchGate. Siehe insbes. Extended Data Fig. 2: Histogram depicting the replication of Zamilon and Sputnik 3 DNA in Mimiviridae after its phylogenetic classification into lineages A, B and C.
- ↑ Ewen Callaway: CRISPR-like ‘immune’ system discovered in giant virus. In: Nature: News, 29. Juni 2016; doi:10.1038/nature.2016.19462.
Auf dieser Seite verwendete Medien
Autor/Urheber: Christoph M. Deeg, Cheryl-Emiliane T. Chow, Curtis A. Suttle, Lizenz: CC BY-SA 4.0
Ultrastructure of Bodo saltans virus (BsV) particles and replication. (A) Mature BsV virion: DNA containing core is surrounded by two putative membranous layers. The capsid consists of at least two proteinaceous layers. The bright halo hints to the presence of short (~40 nm) fibers as observed in Acantha polyphaga memivirus (ApMV). The top vertex of the virion contains a possible stargate structure (Scale bar = 100 nm).
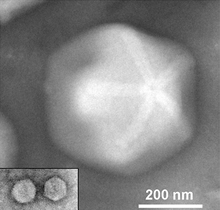
Autor/Urheber: Sarah Duponchel and Matthias G. Fischer, Lizenz: CC BY-SA 4.0
Electron microscope images depicting capsids shape of the giant virus Megavirus vitis (with a visible stargate structure) and its associated virophage Zamilon vitis (inset).
Negative stain EM courtesy of C. Abergel, Aix-Marseille Université, France.
Foto do Tupanvirus
Autor/Urheber: Haruna Takahashi, Sho Fukaya,Chihong Song, Kazuyoshi Murata, Masaharu Takemura, Lizenz: CC BY 4.0
Molecular phylogenetic analysis of family Mimiviridae based on the concatenated NCLDV core genes, consisting of B family DNA polymerase, major capsid protein, D5-like ATPase, mRNA-packaging enzyme, and virion-packaging ATPase. Caption updated on base of ICTV MSL #38.



